聚合酶链式反应(PCR)是分子生物学领域最重要的研究工具之一。在PCR 技术中,DNA聚合酶起着关键性作用[1]。PCR 反应扩增DNA 是在高温条件下进行的,因此需要一种热稳定的酶。Taq DNA 聚合酶最初是从嗜热杆菌(Thrmus aquaticus)中分离纯化得到的热稳定性DNA 聚合酶,是PCR 反应中最常用的一种DNA 聚合酶[2]。而目前大多数实验室主要依赖公司商品化的Taq DNA 聚合酶[3-4]。利用重组大肠杆菌生产 Taq DNA 聚合酶不但可以解决原始菌株产量低,而且可以通过简化纯化步骤,缩短工艺生产流程,为大规模生产Taq DNA 聚合酶打下基础。
由于Taq DNA 聚合酶在PCR 中的广泛应用,有关该酶的制备技术一直在不断地优化提高[5]。为了不断提高该酶的表达量、酶活力和特异性,很多研究主要是针对酶基因本身,表达载体的构建,分离纯化方法。但是对于重组耐热Taq DNA聚合酶在大肠杆菌中发酵生产的最佳表达条件的研究鲜有报道[6]。本文在前期研究的基础上,考察重组大肠杆菌生产Taq DNA聚合酶发酵工艺条件。研究通过单因素以及响应面法优化重组大肠杆菌生产Taq DNA 聚合酶的发酵工艺,进而提高Taq DNA 聚合酶的产量,为Taq DNA 聚合酶的工业化生产提供一定的指导。
1 材料与方法
1.1 材料
E.coli BL21(DE3)/pET28a(+)-Taq:华中农业大学国家微生物重点实验室提供,菌种保存于-80 ℃冰箱中;卡那霉素(Kan)、异丙基-β-D-硫代半乳糖苷(IPTG)、Taq DNA 聚合酶、DNA Marker 等生物化学试剂:上海如吉生物科技发展有限公司。
实验中所用培养基主要有LB 液体培养基和LB 固体培养基及大肠杆菌发酵培养基。Kan 用无菌水配制,50 mg/mL 的母液放置4 ℃保存添加抗生素时,须在培养基冷却至室温时再添加适量的抗生素,避免抗生素在高温时失效。
LB 固体培养基配方:蛋白胨10 g/L、酵母粉5 g/L、NaCl 10 g/L、琼脂15 g/L、1 000 mL 蒸馏水,调节pH 值至7.0~7.5。
LB 液体培养基配方:蛋白胨10 g/L、酵母粉5 g/L、NaCl 10 g/L、1 000 mL 蒸馏水,调节pH值至7.0~7.5。
1.2 仪器与设备
PHM100 全自动手动高温蒸汽灭菌器:武汉集思仪器设备有限公司;SW-CJ-1D 型超净工作台:力辰科技有限公司;HT-211A 型摇床:上海赫田科学仪器有限公司;DNP-9082 型恒温培养箱:宁波江南仪器厂;TC1000-G PCR 仪:北京大龙DLAB。
1.3 实验方法
1.3.1 培养与发酵条件
挑取冻存于-80 ℃环境的E. coli BL21(DE3)/pET28a(+)-Taq 工程菌,接到5 mL 含 Kan 抗性的培养液里37 ℃、180 rpm 振荡一夜。
接种 100 μL 活化的 E.coli BL21(DE3)/pET28a(+)-Taq 于10 mL 含有卡拉霉素(10 μg/mL)的LB 培养液中,37 ℃,250 r/min 培养过夜;次日,取培养过夜的菌液10 mL,接种于1 L 含有卡拉霉素(10 μg/mL)的LB 培养液中,250 r/min培养至对数生长中期(OD600=0.5~0.6);向培养液中加入诱导物IPTG,诱导培养一定时间[7]。
1.3.2 样品的提取
将诱导菌液4 ℃,12 000 r/min 离心5 min,弃去上清液,沉淀重悬于0.01 mol/L PBS(pH 8.0)中;超声波冰浴破壁,破碎产物经4 ℃,12 000 r/min离心15 min 后取上清液;0.45 μm 微孔滤膜过滤上清液,Ni-NTA 亲和层析,洗脱用含有500 mmol/L咪唑的0.01 mol/L PBS(pH 8.0)洗脱,得到重组蛋白溶液[7]。
1.3.3 酶含量的测定——考马斯亮蓝结合法
标准试剂的配制(考马斯亮蓝G-250)[8]:精确称取100 mg 考马斯亮蓝G-250,溶于95%的乙醇中,与100 mL 85% W/V 的磷酸混合,用0.01% M pH 7.0 磷酸缓冲液定溶到1 L,棕色瓶,4 ℃保存。并配置0.01% NaCl 溶液。利用标准曲线测定样品的酶蛋白浓度。为了比较酶制剂的纯度和活力的高低,常常采用比活力这个参数。酶的比活力是酶纯度的一个指标,是指在特定条件下,单位质量(mg)蛋白质所具有的酶活力单位。
1.3.4 酶活单位测定
以购买的Taq DNA 聚合酶进行梯度稀释:1∶2、1∶5、1∶10、1∶15、1∶20。在PCR 反应中,取相同体积不同稀释度的Taq DNA 聚合酶量进行凝胶电泳作为标准梯度;同时,将自制的酶也采用不同的上样量,进行PCR 扩增,凝胶电泳,在凝胶成像系统仪下观察条带的亮度,确定酶的活力及酶的比活力。
1.3.5 单因素实验
采用单因素实验依次对发酵温度、pH、IPTG诱导剂用量、诱导时间、接种量等发酵工艺参数进行逐一优化[9-10]。每组实验重复3 次,对其代谢产物进行含量和酶活力测定,优化能产生Taq DNA 聚合酶含量最大的单因素发酵温度、pH、IPTG 诱导剂用量、诱导时间、接种量[11]。
1.3.6 响应面分析法优化发酵培养基及发酵条件
根据1.2.5 单因素实验结果,以筛选出IPTG 诱导剂用量、诱导时间和接种量作为实验因素[12-13],以Taq DNA 聚合酶含量为响应值,采用响应面实验设计Design-Expert 12 软件的Box-Behnken 设计三因素三水平的实验,并利用响应面法对菌株E. coli BL21(DE3)/pET28a(+)-Taq 最佳发酵工艺进行优化。
1.4 模型的验证实验
将优化后的最佳预测值进行发酵实验,重复3 次,取平均值。比较预测值和实验所得到的实际值,进一步验证响应面法所得到的预测值,来确定响应面法优化的发酵工艺是否准确可靠,是否具有实际的应用价值。
2 结果与讨论
2.1 制备酶蛋白标准曲线
以酶蛋白浓度为横坐标,A595 为纵坐标,得到酶蛋白浓度C 和吸光值A595 线性回归曲线,其方程为y=13.04x-0.000 9,R2=0.999 3,方程式中x 为酶蛋白质浓度,y 为吸光值。酶蛋白标准曲线见图1。
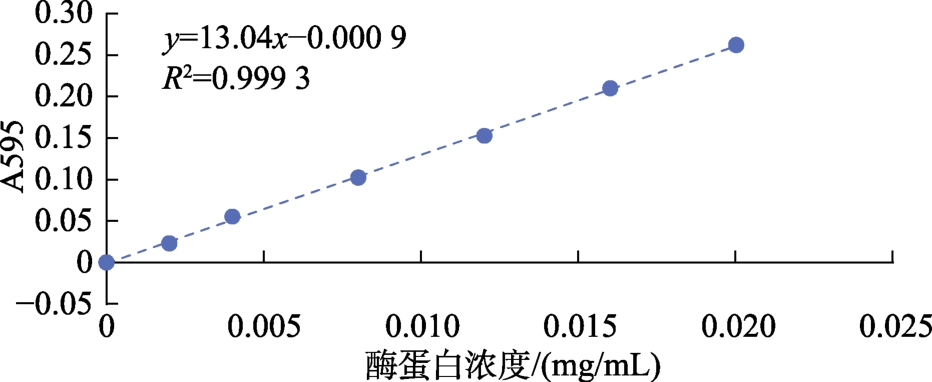
图1 酶蛋白标准曲线
Fig.1 Standard curve of enzyme protein
2.2 单因素实验结果
2.2.1 发酵温度对Taq DNA 聚合酶产量和酶比活力的影响
其它发酵条件保持不变,只改变温度,发酵后测Taq DNA 聚合酶产量和酶比活力。结果如图2 所示。
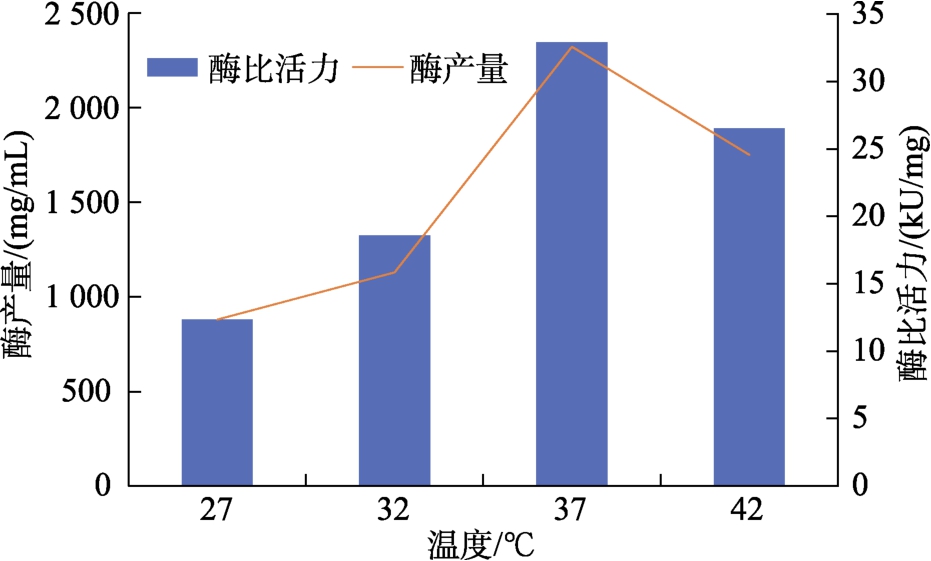
图2 温度对Taq DNA 聚合酶产量和酶比活力的影响
Fig.2 Effect of standard curve temperature of enzyme protein on the yield and specific activity of Taq DNA polymerase
从图2 可以看出,在27~42 ℃发酵条件下,菌体的酶产量和酶比活力随着温度升高而逐渐升高,但是当温度超过37 ℃后,随着温度的升高,酶产量和酶比活力反而下降。可能原因是菌体的生长和产物的合成都是在酶的催化下进行的,而温度是保证酶活性的重要因素,当温度为37 ℃时,Taq DNA 聚合酶产量和酶比活力最大,故为最适发酵温度。
2.2.2 pH 对Taq DNA 聚合酶产量和酶比活力的影响
其它发酵条件保持不变,只改变pH,发酵后测Taq DNA 聚合酶产量和酶比活力。结果如图3所示。
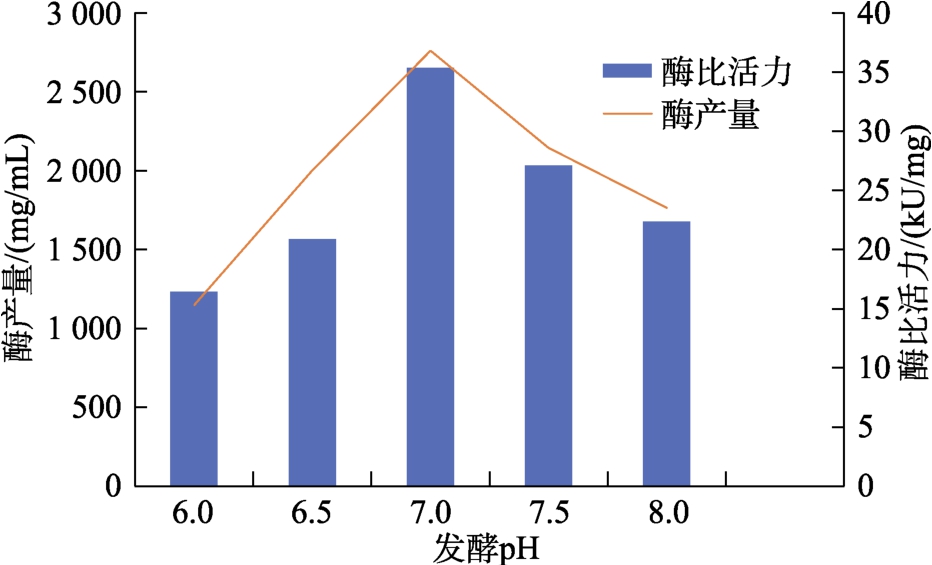
图3 pH 值对Taq DNA 聚合酶产量和酶比活力的影响
Fig.3 Effect of pH on the yield and specific activity of Taq DNA polymerase
从图3 可以看出,当pH 值为6.0~7.0 时,菌体的酶产量和酶比活力明显增加,但是当pH 超过7.0 后,随着温度的升高,酶产量和酶比活力反而下降。可能原因是酸性和碱性环境对菌体生长和酶的合成产生了抑制作用,导致酶比活力下降。故选择最适发酵pH 为7.0。
2.2.3 IPTG 诱导剂用量对Taq DNA 聚合酶产量和酶比活力的影响
其它发酵条件保持不变,只改变IPTG 诱导剂用量,发酵后测Taq DNA 聚合酶产量和酶比活力。结果如图4 所示。
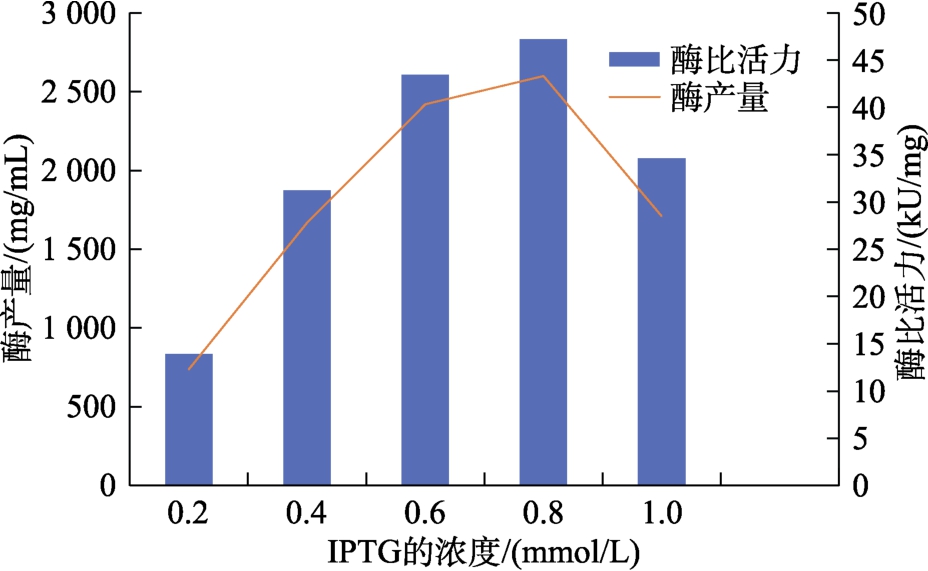
图4 IPTG 浓度对Taq DNA 聚合酶产量和酶比活力的影响
Fig.4 Effect of IPTG concentration on Taq DNA polymerase yield and specific activity
从图4 可以看出,诱导剂IPTG 浓度对Taq DNA 聚合酶产量和酶比活力的影响显著,当IPTG浓度小于0.8 mmol/L 时,随着IPTG 浓度的增加,酶产量和比活力增加,当IPTG 浓度大于0.8 mmol/L时,随着IPTG 浓度的增加,酶产量和酶比活力反而减小。可能原因是IPTG 浓度过低,则达不到与lac 阻遏蛋白结合的饱和浓度,从而不利于诱导蛋白表达;另一方面,过高浓度的IPTG 会促进不溶性的包涵体的形成,导致酶产量和酶比活力下降。所以最佳诱导剂浓度为IPTG(0.8%)。
2.2.4 诱导时间对Taq DNA 聚合酶产量和酶比活力的影响
其它发酵条件保持不变,只改变诱导时间,发酵后测Taq DNA 聚合酶产量和酶比活力。结果如图5 所示。
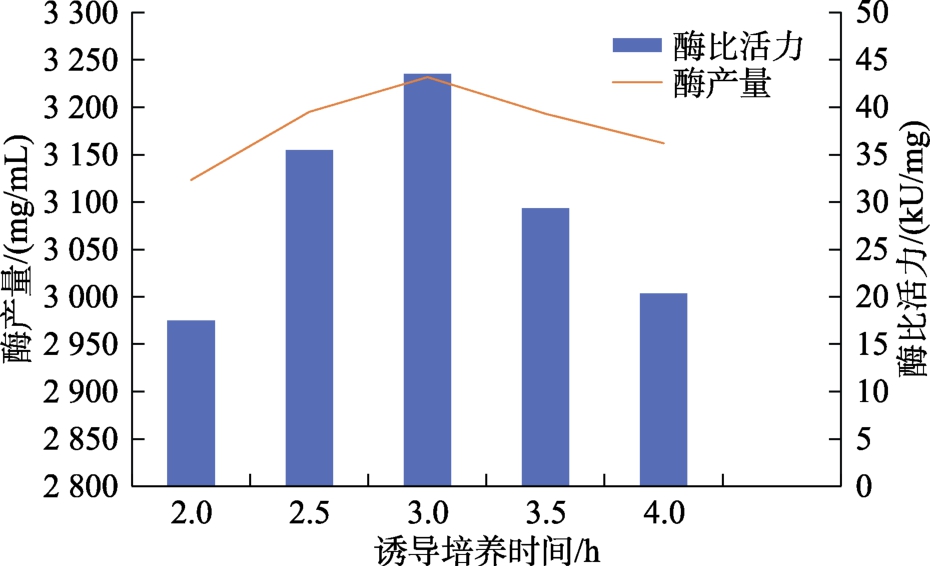
图5 诱导培养时间对Taq DNA 聚合酶产量和酶比活力的影响
Fig.5 Effect of induction culture time on Taq DNA polymerase yield and specific activity
由图5 可以看出,由于菌体大量表达随着诱导时间的增加酶比活力增加,当诱导时间达到3 h,菌体酶产量达到最大,酶比活力达到最大,随着溶氧的下降和代谢产物的积累,菌体表达能力下降,酶比活力也下降。所以最佳诱导培养时间为3 h。
2.2.5 接种量对Taq DNA 聚合酶产量和酶比活力的影响
其它发酵条件保持不变,只改变接种量,发酵后测Taq DNA 聚合酶产量和酶比活力。结果如图6 所示。
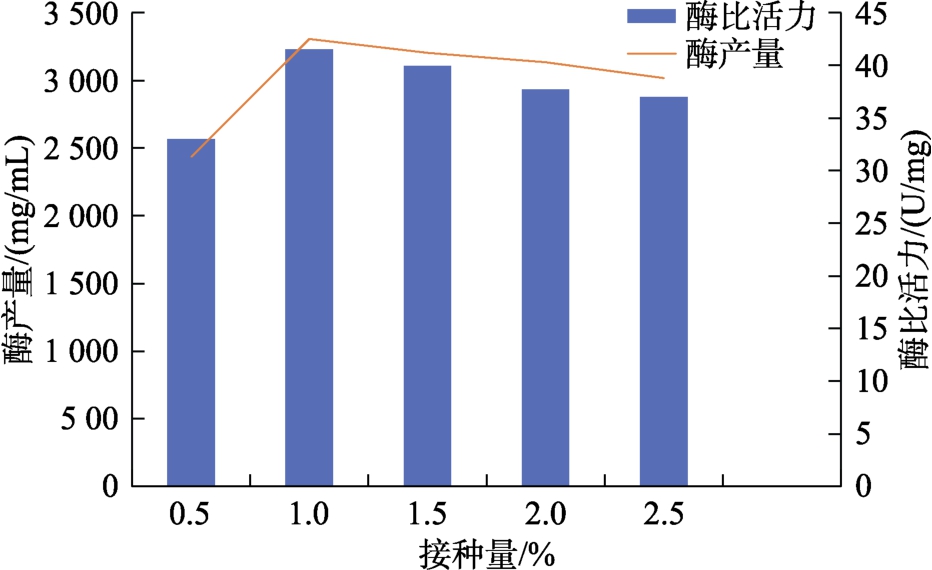
图6 接种量对Taq DNA 聚合酶产量和酶比活力的影响
Fig.6 Effect of inoculation amount on Taq DNA polymerase yield and specific activity
由图6 可以看出,接种量从0.5%增加之1%时,Taq DNA 聚合酶产量和酶比活力随之增加;1%的接种量时,Taq DNA 聚合酶产量和酶比活力最高;接种量从1%增加至2.5%时,Taq DNA 聚合酶产量和酶比活力反而降低。分析原因,接种量低于1%时,菌种量过小,不利于产酶,而接种量超过1%是,菌种量过大,可能会引起菌种生长过快,发酵液黏度增加过大从而导致溶氧不足,影响了酶的表达和酶比活力。所以最佳接种量为1%。
2.3 响应面分析法优化发酵条件的最佳水平
2.3.1 因素水平的筛选
通过对不同因素实验做方差分析,如表1。
表1 实验结果方差分析表
Table 1 Results of variance analysis
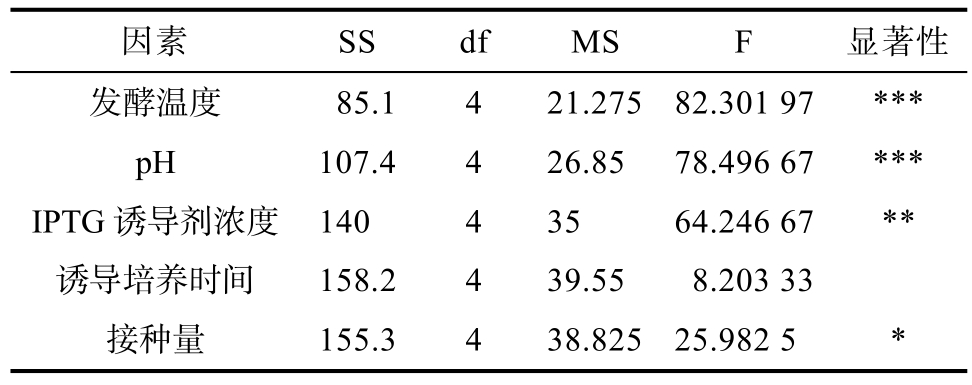
注:(*)表示P<0.05,有差异;(**)表示P<0.01,有显著性差异;(***)表示P<0.001,有极显著性差异。
Note: (*) indicates P<0.05, with differences; (**) indicates P<0.01, with significant differences; (***) indicates P<0.001, with extremely significant differences.
因素 SS df MS F 显著性发酵温度 85.14 21.275 82.301 97***pH 107.44 26.85 78.496 67***IPTG 诱导剂浓度 140 4 35 64.246 67**诱导培养时间 158.24 39.55 8.203 33接种量 155.34 38.825 25.982 5*
通过对发酵温度、pH、IPTG 诱导剂浓度、诱导培养时间、接种量等单因素实验做方差分析(如表1),从分析结果可以看出发酵温度、pH、IPTG诱导剂浓度,对该基因工程菌发酵产Taq DNA 聚合酶比活力有显著影响,而诱导培养时间、接种量对产Taq DNA 聚合酶比活力影响不显著。故选择发酵温度、pH、IPTG 诱导剂浓度作为响应面的三个因素。
2.3.2 实验设计结果
依据单因素结果,选择发酵温度、pH、IPTG诱导剂浓度作为响应面的三个因素,Taq DNA 聚合酶比活力(kU/mg)作为响应值,设计中心组实验,三因素三水平共17 个实验点的响应面分析实验,因素水平选取见表2,实验方案和结果见表3。
表2 三因素三水平取值表
Table 2 Three factors and three levels value table

水平1 0 -1因素 A 温度/℃ 32 37 42 B 发酵pH 6.5 7 7.5 C IPTG 诱导剂浓度/(mmol/L) 0.6 0.8 1.0
表3 实验设计和结果
Table 3 Test design and results

实验号A B C 酶比活力Y 1 37 7 0.8 45.7 2 37 7 0.8 45.6 3 42 6.5 0.8 38.5 4 42 7.5 0.8 35.2 5 37 7 0.8 45.7 6 37 7 0.8 45.8 7 37 6.5 0.6 35.3 8 37 7.5 0.6 30.2 9 32 7.5 0.8 38.3 10 37 7 0.8 45.8 11 42 7 1 26.2 12 42 7 0.6 38.6 13 32 7 1 27.3 14 32 7 0.6 38.9 15 37 7.5 1 33.5 16 37 6.5 1 28.7 17 32 6.5 0.8 36.6
以PCR 反应测定的Taq DNA 聚合酶的比活力(kU/mg)为应值(Y),实验设计结果见表3。
利用Design-Expert12 软件对表3 中的实验数据进行二次多项回归拟合,可以获得Taq DNA 聚合酶比活力对发酵温度、pH、IPTG 诱导剂浓度的二次多项回归方程:y=45.72-0.312 5 A-0.237 5B-0.900 0C-1.25 AB-0.175 0 AC+2.48 BC-6.36 A2-2.21 B2-11.58 C2
2.3.3 模型建立及显著性检验
实验结果方差分析结果见表4。
表4 实验结果方差分析
Table 4 Results of variance analysis
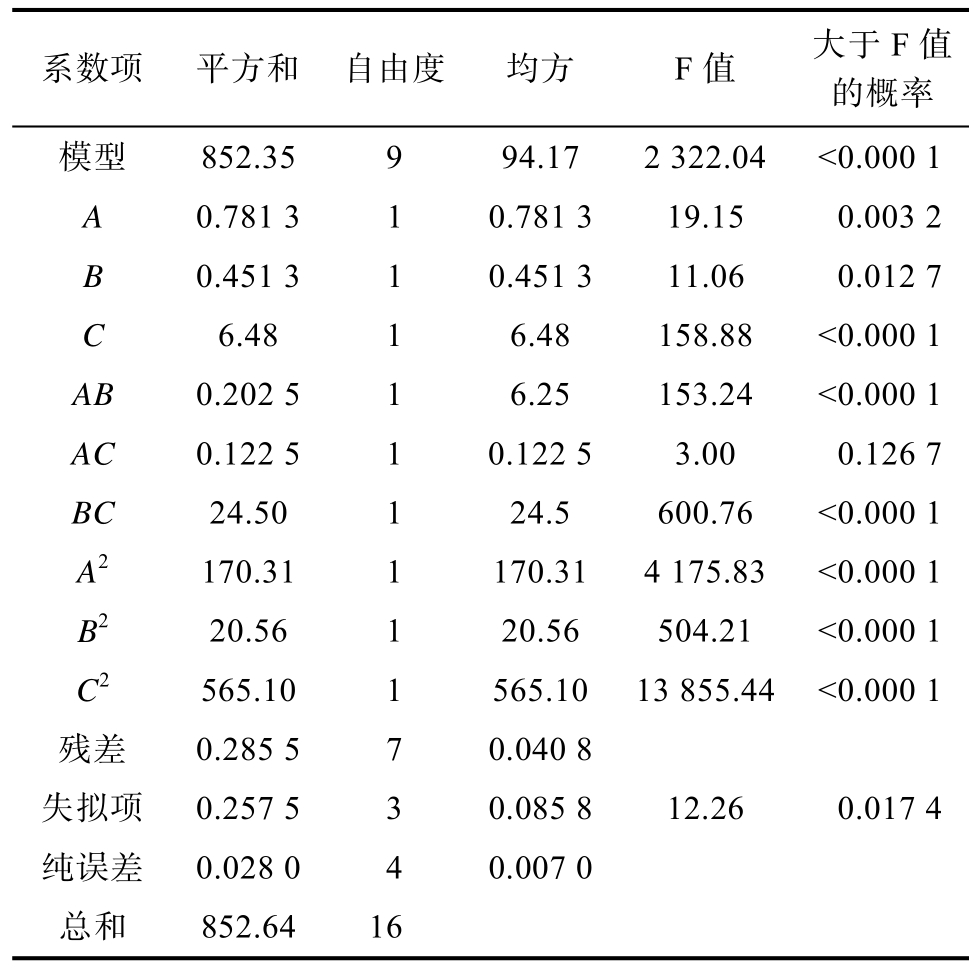
系数项 平方和 自由度 均方 F 值 大于F 值的概率模型 852.35 9 94.17 2 322.04 <0.000 1 A 0.781 3 1 0.781 3 19.15 0.003 2 B 0.451 3 1 0.451 3 11.06 0.012 7 C 6.48 1 6.48 158.88 <0.000 1 AB 0.202 5 1 6.25 153.24 <0.000 1 AC 0.122 5 1 0.122 5 3.00 0.126 7 BC 24.50 1 24.5 600.76 <0.000 1 A2 170.31 1 170.31 4 175.83 <0.000 1 B2 20.56 1 20.56 504.21 <0.000 1 C2 565.10 1 565.10 13 855.44 <0.000 1残差 0.285 5 7 0.040 8失拟项 0.257 5 3 0.085 8 12.26 0.017 4纯误差 0.028 0 4 0.007 0总和 852.64 16
通过对回归分析结果数据进行分析,一次项A(P=0.003 2)极显著,B(P=0.012 7)显著,C(P<0.000 1)极显著,二次项A2(P<0.000 1),B2(P<0.000 1),C2(P<0.000 1)极显著,交互项AB(P<0.000 1),BC(P<0.000 1)极显著,模型回归效果(P=0.000 2<0.005),效果极显著,相关系数R2=0.999 2,说明该模型拟合程度良好,实验误差小,预测值与实测值之间有高度相关性(R2=0.995 1),说明该模型是合适的,适用用来预测和分析重组大肠杆菌生产Taq DNA聚合酶比活力。
2.3.4 响应面分析与优化
固定1 个因素的最有利条件,建立其余2 个因素与响应值关系的三维坐标图。响应面分析结果见图7~9。图7~9 直观反映了各因素对响应值的影响关系:IPTG 浓度影响最大,发酵温度影响也较大,表现为陡峭曲线,pH 影响较小,表现为平滑曲线,pH 值的改变对响应值影响较小。两因素交互作用显著性:发酵温度、IPTG 浓度的交互作用极显著,pH、IPTG 浓度的交互作用极显著,发酵温度、pH 的交互作用不显著。初始值IPTG浓度的一次项系数较大,F 值和P 值检验均反映出IPTG 浓度影响极为显著。说明在重组大肠杆菌生产Taq DNA 聚合酶的过程中IPTG 浓度是限制性因素。响应面显示:IPTG 浓度较小的波动均能引起酶比活力的显著变化。
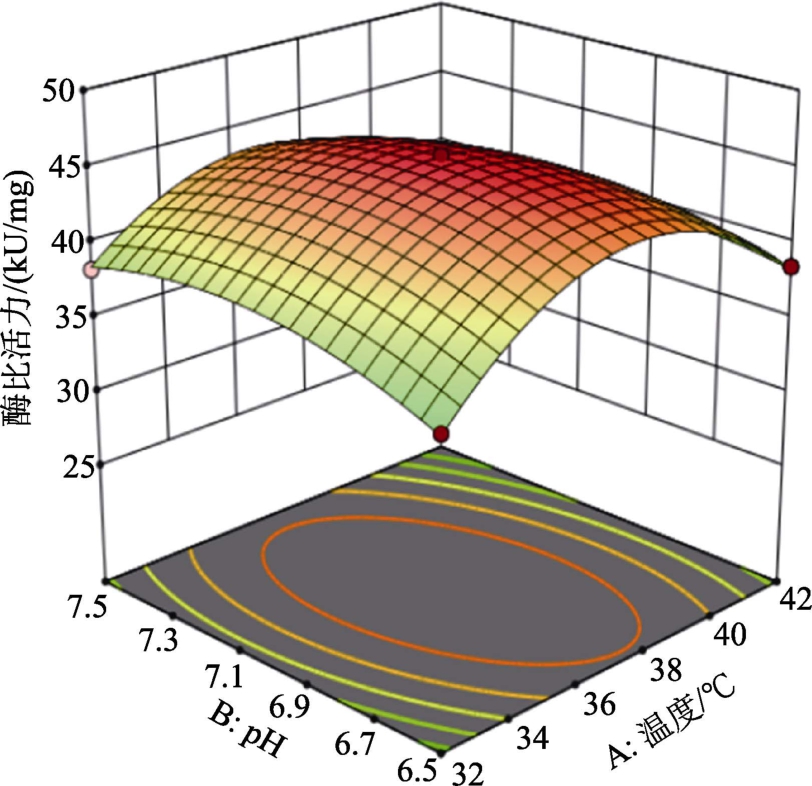
图7 酶比活力与发酵温度、pH 的响应面图
Fig.7 Response surface of enzyme specific activity

图8 酶比活力与pH、IPTG 浓度的响应面
Fig.8 Response surface of enzyme specific to fermentation temperature and pH activity and pH, IPTG concentration
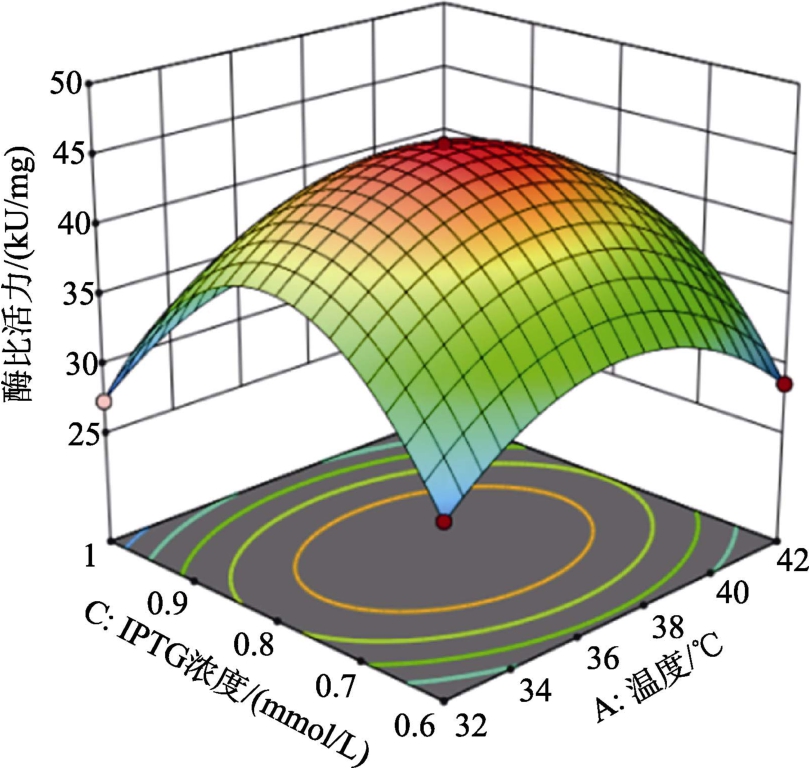
图9 酶比活力与发酵温度、IPTG 浓度的响应面图
Fig.9 Response surface of enzyme specific activity to fermentation temperature and IPTG concentration
通过单因素和交互影响分析以及二次多项式回归模型方程被确定,对方程回归分析,得到重组大肠杆菌生产Taq DNA 聚合酶最佳发酵工艺条件为:发酵温度为37.209 ℃,pH 值为7.468,IPTG诱导剂浓度为0.800 mmol/L,最大酶比活力为43.489 kU/mg。
为了验证模型的准确性和有效性,在最优条件下对重组大肠杆菌生产Taq DNA 聚合酶,重复6 次实验,实验结果分别为:43.186、43.690、45.122、42.768、43.563、44.600 kU/mg,求其平均值为(43.822±0.878)kU/mg,与预测值正偏差0.76%。十分接近理论值。
3 结论
在Taq DNA 聚合酶的表达制备研究实验中Engelke 等[14]制备的Taq DNA 聚合酶的比活力为5 300 U/mg,肖朝文等[15]制备的Taq DNA 聚合酶的比活力为2 651 U/mg,贾平等[16]制备的Taq DNA 聚合酶的比活力为41.667 kU/mg。本实验所用菌株E.coli BL21(DE3)/pET28a(+)-Taq 能高效稳定的表达Taq DNA 聚合酶。通过研究了发酵温度、pH、IPTG 诱导剂用量、诱导时间、接种量等参数对重组大肠杆菌生产Taq DNA 聚合酶的影响。利用Design-Expert12 软件,筛选并确定了发酵生产Taq DNA 聚合酶的主要因素为:IPTG 诱导剂浓度、发酵温度、pH。且显著性为:IPTG诱导剂浓度>发酵温度>pH 再通过响应面实验分析法对发酵工艺进行优化,取得较好的效果。最终确定重组大肠杆菌生产Taq DNA 聚合酶的最佳发酵工艺参数为:发酵温度37.2 ℃,pH 为7.5,IPTG 诱导剂浓度为0.800 mmol/L,Taq DNA 聚合酶比活力为43.822 kU/mg,对酶比活力有明显提高。以此发酵工艺条件为指导,后期可以尝试在发酵罐中进行发酵工艺优化研究,以期望为Taq DNA 聚合酶大批量发酵生产提供指导。
[1] POROWIŃSKA D, WUJAK M, ROSZEK K, et al. Prokaryotic expression systems[J]. Postepy higieny i medycyny doswiadczalnej,2013, 5(24): 29-119.
[2] LIN H L, LI W G, GUO C H, et al. Advances in the study of directed evolution for cellulases[J]. Frontiers of Environmental Science and Engineering in China, 2011, 5(4): 519-525.
[3] 东北制药集团辽宁生物医药有限公司. 热启动Taq DNA 聚合酶及其制备方法与应用: CN201711227574.2[P]. 2021-01-12.Northeast Pharmaceutical Group Liaoning Biopharmaceutical Co., Ltd. Hot start Taq DNA polymerase and its preparation method and application: CN201711227574.2[P]. 2021-01-12.
[4] 天津强微特生物科技有限公司. 一种突变Taq DNA 聚合酶及其纯化方法:CN201611255187.5[P]. 2020-07-14.Tianjin Qiangweite Biotechnology Co., Ltd. A mutant Taq DNA polymerase and its purification method: CN201611255187.5[P].2020-07-14.
[5] 鞠守勇. Taq DNA 聚合酶表达载体的构建及表达条件优化[J].武汉职业技术学院学报, 2022, 21(5):106-109.JU S Y. Construction of Taq DNA polymerase expression vector and optimization of expression conditions[J]. Journal of Wuhan Polytechnic, 2022, 21(5): 106-109.
[6] 孙梅, 葛丽娜, 谢龙, 等. 不同DNA 聚合酶突变体与野生型检测性能的比较分析[J]. 分子诊断与治疗杂志, 2022, 4(2):97-200.SUN M, GE L N, XIE L, et al. Comparative analysis of different DNA polymerase mutants and wild-type detection performance[J].Journal of Molecular Diagnosis and Therapy, 2022, 4(2): 97-200.
[7] 丁燕华. Taq DNA 聚合酶耐热域的克隆及分析[D]. 浙江: 浙江师范大学, 2011.DING Y H. Cloning and analysis of the thermostable domain of Taq DNA polymerase[D]. Zhejiang: Zhejiang Normal University,2011.
[8] 李舒月, 曹春雨, 张浩, 等. 腺相关病毒衣壳保守区多价抗原肽的原核表达及多克隆抗体制备[J]. 南方医科大学学报,2022, 42(6): 944-948.LI S Y, CAO C Y, ZHANG H, et al. Prokaryotic expression of a recombinant protein of adeno-associated virus capsid conserved regions and preparation of its polyclonal antibody[J]. Journal of Southern Medical University, 2022, 42 (6): 944-948.
[9] 高书锋, 孔利华, 雷平, 等. 枯草芽孢杆菌JY24 菌株产β-D-葡萄糖醛酸苷酶发酵工艺优化及催化转化[J]. 生物学杂志,2021, 38(5): 110-115.GAO S F, KONG L H, LEI P, et al. Optimization of the fermentation process and preliminary study of catalytic conversion of β-D-glucuronidase from Bacillus subtilis JY24 strain[J]. Journal of Biology, 2021, 38(5): 110-115.
[10] 查凡, 周柱, 张琴, 等. 克雷伯氏菌乙醛脱氢酶基因过表达及产乙醇发酵工艺优化[J]. 中国酿造, 2022, 41(11): 66-72.ZHA F, ZHOU Z, ZHANG Q, et al. Overexpression of acetaldehyde dehydrogenase gene in Klebsiella sp. and optimization for fermentation process of ethanol production[J]. China Brewing, 2022, 41(11):66-72.
[11] 梁文凤, 侯媛媛, 郭晓磊, 等. 重组左聚糖蔗糖酶生产左聚糖工艺优化[J]. 食品研究与开发, 2023, 44(2): 80-86.LIANG W F, HOU Y Y, GUO X L, et al. Process optimization of levan production by recombinant levansucrase[J]. Food Research and Development, 2023, 44(2): 80-86.
[12] 陈柏林, 汪贵斌, 郭起荣, 等. 响应面法优化白果酒酶解及发酵工艺研究[J]. 南京林业大学学报(自然科学版), 2022, 46(5):135-142.CHEN B L, WANG G B, GUO Q R, et al. Optimization of enzymatic hydrolysis and fermentation conditions of Ginkgo biloba wine using response surface methodology[J]. Journal of Nanjing Forestry University (Natural Science Edition), 2022, 46(5): 135-142.
[13] 名晓东, 郭自涛, 陈剑雄, 等. 重组大肠杆菌产卤代烷脱卤酶的发酵条件优化[J]. 食品与发酵工业, 2022, 48(13): 16-24.MING X D, GUO Z T, CHEN J X, et al. Optimization of fermentation conditions of recombinant Escherichia coli. expressing haloalkane dehalogenase[J]. Food and Fermentation Industries,2022, 48(13): 16-24.
[14] ENGELKE D R, KRIKOS A, BRUCK M E, et al. Purification of Thermus aquaticus DNA polymerase expressed in Escherichia coli[J]. Anal Biochem, 1990, 191(2): 396-400.
[15] 肖朝文. Taq DNA 聚合酶制备技术的优化[D]. 四川农业大学,2005.XIAO C W. Optimization of Taq DNA polymerase preparation technology[D]. Sichuan Agricultural University, 2005.
[16] 贾平. 高纯度Taq DNA 聚合酶制备技术研究[D]. 南京理工大学, 2013.JIA P. Research on the preparation technology of high-purity Taq DNA polymerase[D]. Nanjing University of Science and Technology, 2013.
备注:本文的彩色图表可从本刊官网(http//lyspkj.ijournal.cn)、中国知网、万方、维普、超星等数据库下载获取。