全谷物是健康膳食的重要组成部分,富含膳食纤维和多种生物活性成分。全谷物健康益处很多,包括降低心脏病、脑卒中、II 型糖尿病、结直肠癌风险,减少肥胖,减少炎症、延缓衰老等[1-5]。全谷物健康促进作用已达成国际共识,最新版《中国居民膳食指南(2022)》也增加了每天摄入全谷物和杂豆类50~150 g 的内容。全谷物食品还存在口感粗糙、适口性差、稳定性差、不易储藏等问题。研究证明,发酵可明显提高全谷物制品中生物活性物质的种类和含量,显著增强功能活性,并提升全谷物制品的口感和风味[6-11]。发酵技术在全谷物加工中具有很好的应用前景。
甜醅正是我国传统发酵全谷物食品和特色饮品,主要由燕麦、青稞、糙米制作,历史渊源流长,深受人民喜爱[12-13]。甜醅中蕴含丰富的微生物资源,但对于其微生物区系却少有系统报道,亟待研究阐明。甜醅的研究综合了全谷物食品、发酵技术和微生物资源等方面,对于研究开发全谷物发酵食品具有很好的借鉴作用。
本文以我国典型地区传统甜醅样品为研究对象,利用高通量扩增子测序技术进行甜醅样品细菌微生物区系研究,并解析其细菌群落结构及细菌与甜醅性质关系信息。研究为传统全谷物发酵食品微生物资源发掘、甜醅品质改善及工业化生产等奠定理论基础,也为其他发酵食品微生物研究提供参考。
1 材料与方法
1.1 材料试剂
我国典型地区甜醅样品三种,甜醅样品HN:南方湖南衡阳家庭作坊;HZ:甘肃会洲家庭作坊;LD:北京兰州饭店。
甜醅总基因组利用QIAGEN DNeasymericon Food Kit 试剂盒:美国QIAGEN 公司提取;16S 扩增子文库构建使用NEXTflexTM Rapid DNA-Seq Kit 试剂盒:美国Bio Scientific 公司;甜醅营养化学性质测定使用相应试剂盒测定:北京索莱宝生物科技有限公司。
1.2 仪器与设备
NanoDrop2000 超微量分光光度计:美国Thermo Scientific 公司;V2S02S 微型旋涡仪:德国IKA 公司;SynergvHT 酶标仪:美国BIOTEK公司;ABI GeneAmp® 9700 型PCR 仪:美国ABI公司;EPPENDORF 5810R 台式高速冷冻离心机:德国EPPENDORF 公司;HH-4 数显恒温水浴锅:江苏盛蓝仪器公司;SBA-40E 生物传感分析仪:山东省科学院生物研究所。
1.3 实验方法
1.3.1 甜醅样品基因组的提取
每份甜醅均一化搅拌均匀后称取1 g,参照试剂盒步骤提取甜醅样品的总基因组,每种甜醅样品取三份提取三份基因组作为3 个生物学重复。甜醅DNA 的浓度和纯度利用NanoDrop2000 检测,检测基因组DNA OD260 nm/OD280 nm 值在1.8~2.0 之间,甜醅总DNA 样品置于-80 ℃备用。
1.3.2 细菌16S rRNA 基因PCR 扩增、检测及测序
利用引物799F(AACMGGATTAGATACCCKG)and 1193R(ACGTCATCCCCAC CTTCC)PCR 扩增样品细菌16S rRNA V5-V7 区域[14]。扩增过程为94 ℃预变性5 min,32 个循环包括94 ℃变性30 s,53 ℃退火30 s, 72 ℃延伸 48 s,再72 ℃延伸8 min,4 ℃保存。PCR 反应体系20 μL,包括5×PCR Mix 4 μL,引物799F(10uM)1 μL,引物1193R(10uM)1 μL,DNA 模板10 ng,ddH2O补齐。每种甜醅样品3 个重复。将同样本的PCR产物混匀后通过1.8%琼脂糖凝胶电泳切胶回收PCR 产物并纯化,PCR 回收产物检测定量,文库构建,在上海美吉生物医药科技有限公司进行细菌扩增子测序,测序平台为美国 Illumina 公司Miseq PE300。细菌扩增子的原始数据提交至NCBI SRA 数据库,序列号为PRJNA905189。
1.3.3 生物信息学分析
细菌扩增子原始序列的质控采用 Fastp[15]0.19.6 软件比对,序列拼接利用FLASH[16]1.2.11软件。质控内容包括设置窗口值为50 bp,过滤掉reads 尾部质量值小于20 的碱基;最小重叠长度为10 bp,依据重叠关系,拼接成对reads 成序列;拼接序列重叠区允许的最大错配比率为0.2,去除不符合条件的序列;根据标记序列和引物区分不同样品,并调整好序列方向,标记序列错配数值设为0,最大引物错配数值设为2。通过UPARSE[17]7.1 软件进行可操作分类单元(OTU)聚类,设置相似度为97%[17-18]并剔除嵌合体序列。利用RDP classifier[19] 2.13 对每条序列进行silva138 数据库比对并给出物种分类注释。利用PICRUSt2 2.2.0 [20]进行细菌物种功能预测,利用Lefse (http://huttenhower.sph.harvard.edu/galaxy/ root?tool_id=lefse_upload)筛选出甜醅样本中显著性差异影响的细菌分类单元。
2 结果与讨论
2.1 测序数据概况及甜醅细菌α 多样性
研究完成了甜醅样本的多样性数据测序和分析,共获得优化序列481 313 条,包含181 512 717碱基,平均序列长度377 bp。物种注释结果统计甜醅样品中包括细菌界26 门、55 纲、119 目、193科、314 属、417 种和504 个可操作分类单元(OTU)。甜醅样品细菌α 多样性指数见表1。甜醅样品的Coverage 值都大于0.999,证明测序覆盖率很好,测序深度适合。分别选取Chao1 代表样品丰度(Richness)、Shannon 代表多样性(Diversity)、Simpsoneven 代表均一性(evenness)。甜醅样品中HN 具有最高的丰富度和多样性,甜醅HZ 具有最高的均一性;而甜醅样品LD 丰富度、多样性和均一性值均为最低。
表1 甜醅细菌α 多样性指数表
Table 1 α diversity indices of Tianpei samples
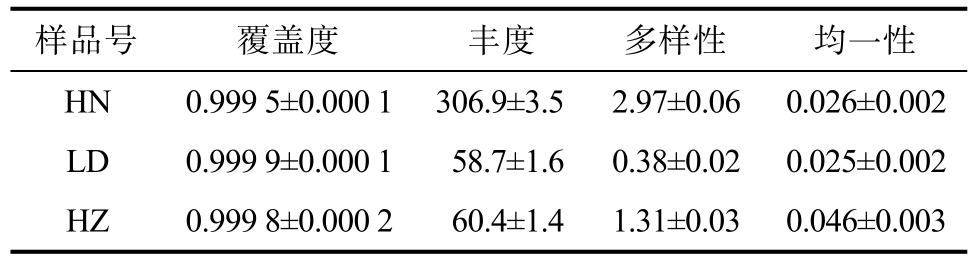
样品号覆盖度 丰度 多样性 均一性HN 0.999 5±0.000 1 306.9±3.5 2.97±0.06 0.026±0.002 LD 0.999 9±0.000 158.7±1.6 0.38±0.02 0.025±0.002 HZ 0.999 8±0.000 260.4±1.4 1.31±0.03 0.046±0.003
2.2 甜醅样品细菌区系组成
在细菌门水平,甜醅样品中有4 个门的丰度大于1%,分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteriota)和拟杆菌门(Bacteroidota)。厚壁菌门(Firmicutes)含量最高(甜醅HN-55.4%,LD-96.3%,HZ-98.4%),其次是变形菌门(Proteobacteria),在甜醅HN 中含量最高达 30.1%(图 1A)。另外,放线菌门(Actinobacteriota)在甜醅HN 中含量11.2%。厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)细菌在多种发酵食品中广泛分布,并作为发酵食品微生物区系的主要细菌类群[21-23]。由于其强大和广泛的适应性,它们也是许多其他环境系统中的优势微生物群[24-25]。放线菌门成员在土壤、海洋等环境分布,还可和植物共生,在自然界的氮素循环中起着一定的作用,其中著名的链霉菌属(Streptomyces)和放线菌属(Actinomyces)等成员能产生多种抗菌素[26]
在属水平,甜醅样品中有15 个属的丰度大于1%,包括魏斯氏属(Weissella)、乳杆菌属(Lactobacillus)、葡萄球菌属(Staphylococcus)、潘隆尼亚碱湖杆菌(Pannonibacter)、芽孢杆菌属(Bacillus)、片球菌属(Pediococcus)、醋酸杆菌(Acetobacter)等(图 1B)。其中,魏斯氏属(Weissella)在甜醅LD 中丰度最高(93.3%),乳杆菌属(Lactobacillus)在甜醅HZ 中丰度最高(86.3%),而甜醅HN 中丰度最高的为Staphylococcus(33.8%)和Weissella(12.8%)(图1B)。三种甜醅样品共享33 个OTUs(图1C),主要隶属于Lactobacillus、Weissella、Staphylococcus、Pediococcus。甜醅LD 中无特有OTU, 甜醅HN 中特有OTU 数目则最多,与上述多样性指数结果相对应。菌群中Weissella、Lactobacillus、Pediococcus 等都属于乳酸菌(LAB),在食品和饮品发酵中具有重要的作用[27-28]。Pannonibacter 成员被报道具有还原Cr(Ⅵ)的特性[29]。本研究中的葡萄球菌为尼泊尔葡萄球菌(Staphylococcus nepalensis),被报道可用于酿造豆瓣,产乳酸,降解苯酚,并且被发现是传统锦州虾酱中产蛋白酶的嗜盐菌[30-31]。其在甜醅中的具体功能作用值得进一步继续研究。

图1 甜醅细菌区系组成图
Fig.1 Diagrams of bacterial composition among sweet fermented oats samples
注:A. Circos 图门水平;B. Circos 图属水平;C. 维恩图.
Note: A. Circos diagram at phylum level; B. Circos diagram at genus level; C. Venn diagram.
2.3 甜醅样品细菌β 多样性和多级物种差异判别分析
主坐标分析(PCoA)在OTU 水平上,显示3种甜醅样品细菌形成各自的聚集群落(图2)。甜醅样品细菌群落差异特性值PC1 为46.36%,PC2 为40.05%。利用ANOSIM 分析,即相似性分析,也证实了3种甜醅样品细菌群落存在差异(P =0.001)。ANOSIM 分析属于非参数检验,经常用来检验组间差异是否显著大于组内差异,进而判断分组是否具有意义。本研究中甜醅样品来自不同地区,不同原料,表现出了不同的细菌多样性及菌落结构。
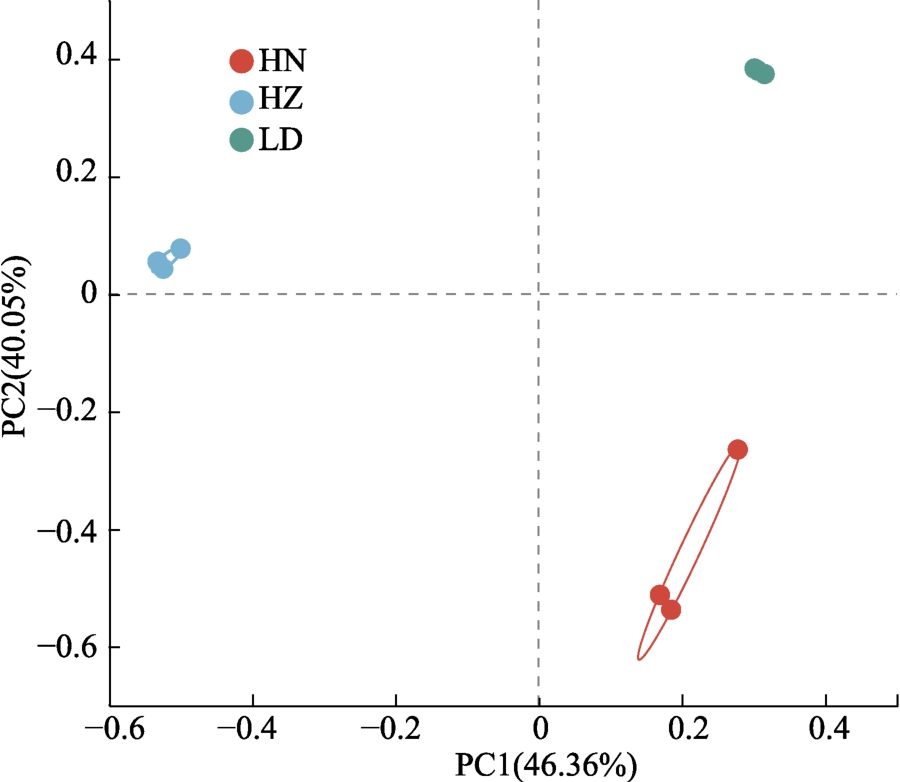
图2 甜醅细菌PCoA 图
Fig.2 PCoA diagram of bacterial community among Tianpei samples
Lefse 分析可发现高维生物标识物并揭示基因组特征,区别不同生物类群。其算法强调统计意义和生物相关性,能够识别不同丰度特征及其关联的类别,根据分类学组成对样本进行线性判别分析(LDA),找出样本之间产生显著性差异影响的分类单元[22]。在我们研究的甜醅样品中,通过Lefse 分析发现了与样品相关的指示或生物标志分类群,细菌标志类群筛选中,以LDA 值>4为标准,不同类群之间存在显著差异(图3)。利用单因素方差分析(One-way ANOVA)检验进行了验证。甜醅样品HN 丰度和多样性最高,细菌标志分类群也最多。在属水平,细菌标志分类群主要包括乳杆菌属(Lactobacillus)、葡萄球菌属( Staphylococcus )、 潘隆尼亚碱湖杆菌(Pannonibacter)、芽孢杆菌属(Bacillus)等。这些细菌标志分类群也包含在甜醅主要细菌组成类群中,提示我们重点关注这些类群。

图3 甜醅样品细菌Lefse 分析图
Fig.3 Lefse diagram of bacterial community among Tianpei samples
注:A. 样品Lefse 图;B. LDA 分布图.
Note: A. Lefse cladogram of different taxa among samples; B. LDA value distribution graph.
2.4 甜醅细菌与甜醅性质关系及甜醅细菌功能预测分析
三种甜醅样品性质见表2,包括乳酸,黄酮、氨基酸、蛋白、淀粉、游离脂肪酸和还原糖含量。通过db-RDA 关联分析研究发现,甜醅细菌组成与甜醅性质显著相关(图4)。利用Spearman 关联分析发现魏斯氏属(Weissella)与氨基酸含量和还原糖含量显著相关、乳杆菌属(Lactobacillus)与蛋白含量显著相关、葡萄球菌属(Staphylococcus)、芽孢杆菌属(Bacillus)和潘隆尼亚碱湖杆菌(Pannonibacter)则与淀粉含量显著相关。目前,全谷物发酵研究中报道了多种乳酸菌发酵促进燕麦、糙米等改性,提升口感和增强风味,提高产品营养价值等[10,32-33]。关于我国传统全谷物发酵食品甜醅微生物区系的研究还很少,特别是微生物与其性质相关性方面的研究[34],今后有待进一步加强。本部分研究为细菌与甜醅理化性质相关性研究及后续甜醅性质改良提供参考。

图4 db-RDA 分析图
Fig.4 db-RDA diagram of bacterial community among Tianpei samples
注:LA—乳酸; F—黄酮; AA—氨基酸;P—蛋白; S—淀粉; FA—游离脂肪酸;RS—还原糖.
Note: LA—lactic acid; F—flavonoid;AA—amino acid; P—protein; S—starch;FA—free fatty acid; RS—reducing sugar.

图5 基于16S rDNA 序列甜醅细菌功能预测热图
Fig.5 Functional analysis heatmap of bacterial community among Tianpei samples based on 16S rDNA
表2 甜醅样品营养化学性质表
Table 2 Chemical properties of Tianpei samples
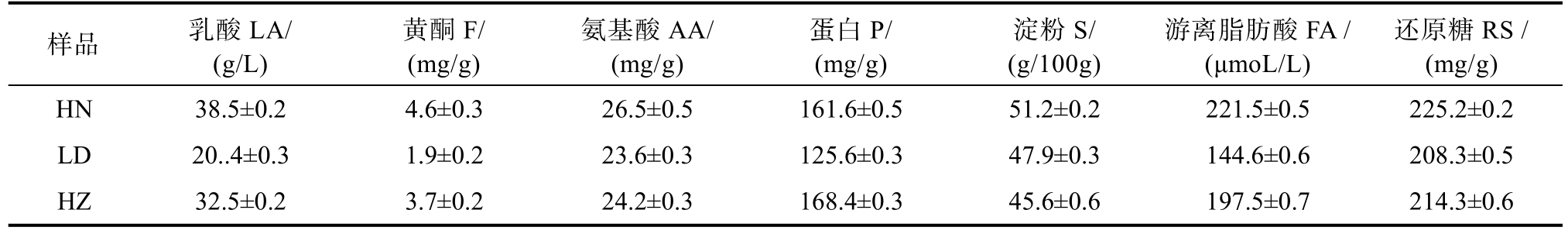
样品 乳酸LA/(g/L)黄酮F/(mg/g)氨基酸AA/(mg/g)蛋白P/(mg/g)淀粉S/(g/100g)游离脂肪酸FA /(μmoL/L)还原糖RS /(mg/g)HN 38.5±0.2 4.6±0.3 26.5±0.5 161.6±0.5 51.2±0.2 221.5±0.5 225.2±0.2 LD 20..4±0.3 1.9±0.2 23.6±0.3 125.6±0.3 47.9±0.3 144.6±0.6 208.3±0.5 HZ 32.5±0.2 3.7±0.2 24.2±0.3 168.4±0.3 45.6±0.6 197.5±0.7 214.3±0.6
利用PICRUSt 基于16S 扩增子测序结果开展细菌功能预测,其通过每个OTU 对应的greengene id,获得OTU 对应的COG 信息和KEGG Ortholog(KO)信息;并计算各COG 和KO 丰度[35]。PICRUSt 可获得代谢通路Pathway 在3 个水平的信息。细菌菌群的COG 功能主要集中在一般功能(General function)、氨基酸转运和代谢(Amino acid transport and metabolism)、碳水化合物转运和代谢(Carbohydrate transport and metabolism)等。在KEGG pathway 水平3 上,细菌功能主要集中在代谢途径、次生代谢物生物合成、氨基酸降解、氨基酸合成、RNA 降解、碳固定、三羧酸循环等。碳水化合物和蛋白等是发酵谷物原料的重要组成成分[1],因而不难理解其中微生物群落的功能与其代谢相关,今后可结合多组学,包括宏转录组,代谢组等进一步研究分析菌群功能。
3 结论
本研究对我国典型地区的发酵全谷物甜醅样品进行了细菌微生物区系研究,解析了甜醅细菌群落结构及细菌与甜醅性质关系信息。研究结果显示,甜醅样品中有4 个门丰度大于1%,并以厚壁菌门(Firmicutes)含量最高。在属水平,以魏斯氏菌(Weissella)、乳杆菌(Lactobacillus)、葡萄球菌(Staphylococcus)等丰度最高。三种甜醅样品的细菌群落组成结构不同,应与其不同来源和不同原料相关。Lefse 分析发现了每种甜醅样品的生物标志分类单元,包括乳杆菌(Lactobacillus)、葡萄球菌(Staphylococcus)等。通过db-RDA 关联研究发现, 甜醅细菌群落与甜醅性质(乳酸,黄酮、氨基酸、蛋白、淀粉、游离脂肪酸和还原糖含量)显著相关。甜醅细菌主要涉及氨基酸转运和代谢及碳水化合物转运和代谢等。下一步研究可深入结合多组学技术,宏转录组、代谢组等研究挖掘微生物在甜醅发酵过程中的功能作用。研究为传统全谷物发酵食品微生物资源发掘、甜醅品质改善及工业化生产等奠定理论基础,也为其他全谷物发酵食品微生物区系研究提供参考。
[1] 谭斌. 全谷物营养健康与加工[M]. 北京; 科学出版社, 2021.TAN B. Whole Grains nutrition, health benefits and processing[M].Beijing: Science Press, 2021.
[2] HAJIHASHEMI P, AZADBAKHT L, HASHEMIPOUR M, et al.Whole grain intake favorably affects blood glucose and serum triacylglycerols in overweight and obese children: a randomized controlled crossover clinical trial[J]. Nutrition, 2021, 87: 11200.
[3] ALLAI F M, AZAD Z, GUL K, et al. Wholegrains: A review on the amino acid profile, mineral content, physicochemical, bioactive composition and health benefits[J]. International Journal of Food Science & Technology, 2022, 57(4): 1849-1865.
[4] XIONG Y, ZHANG P, WARNER R D, et al. Cereal grain-based functional beverages: from cereal grain bioactive phytochemicals to beverage processing technologies, health benefits and product features[J]. Critical Reviews in Food Science and Nutrition,2022, 62(9): 2404-2431.
[5] TAPOLA N, KARVONEN H, NISKANEN L, et al. Glycemic responses of oat bran products in type2 diabetic patients[J].Nutrition Metabolish and Cardiovascular Diseases, 2004, 15(4):255-261.
[6] ZHAO H M, GUO X N, ZHU K X. Impact of solid state fermentation on nutritional, physical and flavor properties of wheat bran[J]. Food Chemistry, 2017, 217: 28-36.
[7] 彭小霞. 全谷物酸牛奶的发酵技术及营养功能研究[D]. 广州:华南理工大学, 2019.PENG X X. Studies on the fermentation technology and nutritional functionality of whole grain yogurt[D]. Guangzhou:South China University of Technology, 2019
[8] ADEBO O A, GABRIELA MEDINA-MEZA I. Impact of fermentation on the phenolic compounds and antioxidant activity of whole cereal grains: A mini review[J]. Molecules, 2020, 25:927.
[9] ZHANG D Q, YE Y J, WANG L Y, et al. Nutrition and sensory evaluation of solid-state fermented brown rice based on cluster and principal component analysis[J]. Foods, 2022, 11: 1560.
[10] MASIÁ C, GEPPEL A, JENSEN P E, et al. Effect of Lactobacillus rhamnosus on physicochemical properties of fermented plantbased raw materials[J]. Foods, 2021, 10(3): 573.
[11] KEWUYEMI Y O, NJOBEH P B, KAYITESI E, et al. Metabolite profile of whole grain ting (a Southern African fermented product)obtained using two strains of Lactobacillus fermentum[J].Journal of Cereal Science, 2020, 95: 103042.
[12] 史晓萌, 陈建国, 梁寒峭, 等. 燕麦发酵前后营养成分及风味物质分析[J]. 食品科技, 2018, 43(5): 169-174.SHI X M, CHEN J G, LIANG H Q, et al. Changes of nutrients and flavor substances before and after oats fermentation[J]. Food Science and Technology, 2018, 43(5): 169-174.
[13] 吴寒, 肖愈, 李伟, 等. 燕麦甜醅发酵过程中生化成分的动态变化[J].食品科学, 2015, 36(13): 114-118.WU H, XIAO Y, LI W, et al. Dynamic changes in biochemical components during fermentation of sweet fermented oat[J]. Food Science, 2015, 36(13): 114-118.
[14] BULGARELLI D, GARRIDO-OTER R, MÜNCH P C, et al.Structure and function of the bacterial root microbiota in wild and domesticated barley[J]. Cell host & microbe, 2015, 17(3):392-403.
[15] CHEN S, ZHOU Y, CHEN Y, et al. Fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890.
[16] MAGOČ T, SALZBERG S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics.2011, 27(21): 2957-2963.
[17] EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10):996-998.
[18] STACKEBRANDT E, GOEBEL B M. Taxonomic Note: A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic Bacteriology, 1994, 44(4):846-849.
[19] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied Environment Microbiology,2007, 73(16): 5261-5267.
[20] DOUGLAS G M, MAFFEI V J, ZANEVELD J R, et al.PICRUSt2 for prediction of metagenome functions[J]. Nature Biotechnology, 2020, 38: 685-688.
[21] WANG Z, XU Z, SUN L, et al. Dynamics of microbial communities, texture and flavor in Suan zuo yu during fermentation[J]. Food Chemistry, 2020, 332: 127364.
[22] LING H Z, SHI H L, CHEN X C, et al. Detection of the microbial diversity and flavour components of northeastern Chinese soybean paste during storage[J]. Food Chemistry, 2021,374: 131686.
[23] TAMANG J P, DAS S, KHARNAIOR P, et al. Shotgun metagenomics of Cheonggukjang, a fermented soybean food of Korea: community structure, predictive functionalities and amino acids profile[J]. Food Research International, 2022, 151:110904.
[24] DELGADO-BAQUERIZO M, OLIVERIO A M, BREWER T E,et al. A global atlas of the dominant bacteria found in soil[J].Science, 2018, 359: 320-325.
[25] WANG H Y, GUO S Y, HUANG M R, et al. Ascomycota has a faster evolutionary rate and higher species diversity than Basidiomycota[J]. Science China-Life Sciences, 2010, 53:1163-1169.
[26] 尚坤, 阎瑾逸, 高君梅, 等. 放线菌来源活性天然产物发现研究进展[J/OL].微生物学杂志, 1-8[2022-09-23]. http://kns.cnki.net/kcms/detail/21.1186.Q.20220922.1128.002.html.SHANG K, YAN J Y, GAO J M, et al. Advances in the discovery of Actinomycetes originated active natural products[J/OL]. Journal of Microbiology, 1-8[2022-09-23]. http://kns.cnki.net/kcms/detail/21.1186.Q.20220922.1128.002.html.
[27] OJETTI V, SAVIANO A, BRIGIDA M, et al. Randomized control trial on the efficacy of Limosilactobacillus reuteri ATCC PTA 4659 in reducing inflammatory markers in acute uncomplicated diverticulitis[J]. European Journal of Gastroenterology &Hepatology, 2022, 34: 496-502.
[28] ZANNINI E, MAUCH A, GALLE S, et al. Barley malt wort fermentation by exopoly-saccharide-forming Weissella cibaria MG 1 for the production of a novel beverage[J]. Journal of Applied Microbiology, 2013, 115: 1379-1387.
[29] 柴立元, 曾娟, 苏艳蓉, 等. 一株Cr(Ⅵ)还原菌的鉴定及其还原特性[J]. 中南大学学报(自然科学版), 2011, 42: 300-304.HAI L Y, ZENG J, SU Y R, et al. Identification and characteristics of a Cr (Ⅵ) reducing strain Pannonibacter sp.[J].Journal of Central South University (Science and Technology),2011, 42: 300-304.
[30] 姜立春, 纪思萌, 王婷, 等. 苯酚降解菌株JY04 的筛选、分离与鉴定[J]. 绵阳师范学院学报, 2017, 36: 43-47.JIANG L, JI S M, WANG T, et al. Screening, isolation and identification of phenol degrading strain JY04[J]. Journal of Mianyang Teachers’College, 2017, 36: 43-47.
[31] 吕欣然, 李莹, 马欢欢, 等. 传统锦州虾酱中产蛋白酶嗜盐菌的分离与鉴定[J].食品工业科技, 2016, 37: 121-125+136.LV X Y, LI Y, MA H H, et al. Isolation and identification of protease-producing halophilic bacteriain traditional Jinzhou shrimp paste[J]. Science and Technology of Food Industry,2016, 37: 121-125+136.
[32] APARICIO-GARCÍA N, MARTÍNEZ-VILLALUENGA C,FRIAS J, et al. Production and characterization of a novel gluten-free fermented beverage based on sprouted oat flour[J].Foods, 2021, 10(1): 139.
[33] ANGELOV A, YANEVA-MARINOVA T, GOTCHEVA V.Oats as a matrix of choice for developing fermented functional beverages[J]. Journal of food science and technology, 2018,55(7): 2351-2360.
[34] 史晓萌, 陈建国, 梁寒峭, 等.燕麦发酵过程中微生物、理化指标及活性成分变化规律[J]. 食品与发酵工业,2018, 44:49-52.SHI X M, CHEN J G, LIANG H Q, et al. Analysis of microorganism,physicochemical parameters and active components during fermentation of oats[J]. Food and Fermentation Industries, 2018,44: 49-52.
[35] LANGILLE M, ZANEVELD J, CAPORASO J G, et al. Predictive functional profiling of microbial communities using 16s rRNA marker gene sequences[J]. Nature Biotechnology 2013,31(9): 814-821.
备注:本文的彩色图表可从本刊官网(http//lyspkj.ijournal.cn)、中国知网、万方、维普、超星等数据库下载获取。文中所有菌属名为拉丁名,均为斜体。