国家统计局关于 2020年粮食产量的公告数据显示,稻谷产量约占我国粮食总产量的三分之一,是主要粮食作物,也是重要的战略储备粮种。相较于小麦和玉米而言,稻谷具有不耐储藏的性质,一般在粮库储备轮换年限为3年[1]。在收购、储藏和加工过程中,外界环境中的微生物会对稻谷的品质带来一定影响[2]。尤其是在粮库储藏时粮仓内的粮堆是一个复杂的生态系统,仓内外环境温湿度的变化,会导致水分迁移而使粮堆局部出现结露的情况,从而为真菌的萌发生长创造有利条件,可能导致储粮风险[3-4]。
早在2001年,Reddy等[5]总结了水稻中报道过的143种不同的真菌,结果表明无论是在田间还是在储藏期,稻谷上都存在大量真菌。储藏期间稻谷的优势真菌主要为曲霉属Aspergillus 物种[6]。包括黄曲霉A. flavus、黑曲霉A. niger、亮白曲霉A. candidus等[7]。曲霉属Aspergillus内不同真菌物种之间,生长条件及产毒特性差异较大,对稻谷的危害程度不同[8],准确鉴定主要优势真菌物种,对于粮食霉变及真菌毒素污染精准防控具有重要意义。
早期粮食上真菌的鉴定多以形态特征鉴定为主,部分种属的鉴定结果可能存在不准确[9]。随着分子生物学的建立与发展,现代分子生物学技术手段被逐渐引入粮食真菌的基因序列分析,可将分离到的真菌鉴定到属和种水平[7,10]。目前主要采用内转录间隔区(Internal Transcribed Spacer,ITS区)作为通用条形码对储藏真菌进行初步鉴定,可将分离株鉴定至属或属内复合群水平。但ITS序列在 Genbank数据库中比对分析获得的菌种鉴定信息具有一定的局限性[11-12],尤其是曲霉属Aspergillus(粮食储藏真菌优势属),ITS序列分析过于保守,不能将所有的物种准确鉴定到单一种[12],因此,需聚合酶Ⅱ第二大亚基基因(RNA polymerase II, RPB2)、β-微管蛋白基因(β-tubulin,TUB)或钙调蛋白基因(Calmodulin, CAM)等多个蛋白编码基因对复合群内菌株进行准确种水平鉴定[13]。本文选择将形态学观察与多基因系统发育分析相结合,对我国13省粮库储藏期间稻谷上优势真菌进行准确鉴定,为粮食上危害真菌的识别和鉴定提供参考。
1 材料和方法
1.1 实验样品
本研究稻谷样品分别来源于黑龙江、吉林、辽宁、宁夏、北京、安徽、湖北、湖南、江西、四川、重庆、福建、广西13个省储存1年的粮仓(高大平房仓)。每仓采样方法为5点3层法,共计15个样品,5个取样点平面位置定为粮仓的东南角、东北角、西南角、西北角以及中央点,3层的纵向位置分别为:上层(距离粮堆表层0.5 m左右)、中层(粮堆正中间,通常距粮堆表层2.5~3 m左右,根据粮仓内粮堆高度而定)和下层(距离粮仓地面0.5 m左右),取样后,将样品放置于无菌采样袋中,于4 ℃条件下保存,实验前将上层、中层、下层样品分别进行混合。
1.2 主要试剂和仪器
高盐察氏琼脂培养基(Salt Czapek Dox Agar,SCA)、察氏琼脂培养基(Capek agar, CA)、马铃薯葡萄糖琼脂培养基(Potato Dextrose Agar,PDA)、麦芽汁琼脂培养基(Malt Extract Agar,MEA):北京陆桥技术股份有限公司;十六烷基三甲基溴化铵(CTAB):宝泰克生物科技公司;2×PCR Master Mix、DL2000 DNA Marker:天根生化科技(北京)有限公司;引物:生工生物工程(上海)股份有限公司;科研级生物显微镜(Axio scopeA1):德国zeiss公司;PCR仪(Tgradient):Biometra公司;凝胶成像分析仪(Gel-Doc2000):Bio-Rad公司。
1.3 实验方法
1.3.1 稻谷上真菌的分离计数和纯化
储藏稻谷真菌的分离主要采用稀释平板法(SCA培养基)。称取 25 g稻谷样品至装有225 mL无菌水的锥形瓶中,振荡30 min,充分洗涤后,制备10倍梯度的真菌孢子稀释液,选择2个适宜的稀释梯度进行接种,每份样品三个平行。接种后及时将15~30 mL冷却至50 ℃左右加了氯霉素的 SCA培养基倒入已加稀释液的无菌培养皿中。待琼脂凝固后,置于 28 ℃恒温培养箱中培养5~8 d。记录稀释倍数和相应真菌菌落数。计数后挑取单菌落边缘菌丝转接3~4次,纯化获得单一菌株。
1.3.2 真菌形态特征鉴定
菌落表观形态观察,选择MEA培养基、CA培养基或PDA培养基用于菌株培养,在25 °C培养箱中培养7 d进行观察。微观形态特征观察使用科研级生物显微镜。详细方法参考Chen等的实验方法[14]。
1.3.3 真菌分子生物学鉴定
使用改良的CTAB(十六烷基三甲基溴化铵)法提取基因组DNA。用于ITS区及TUB基因序列的扩增和测序。菌落数较多的分离株及与其形态近似的菌株扩增 CAM基因和 RPB2基因并测序。ITS区的引物选择ITS4和ITS1;TUB基因扩增选择引物bT2a和bT2b;RPB2基因扩增选择引物RPB2-5F和RPB2-7R;CAM基因扩增选择引物CMD5和CMD6。PCR反应体系为25 μL:引物各 1 μL,模板 DNA 1 μL,2×PCR Master Mix为12.5 μL,ddH2O为9 μL。反应条件参考Chen等的研究方法[14]。
1.4 数据分析
所有数据采用 EXCEL软件进行统计分析。ITS、TUB、CAM 以及 RPB2单基因的序列先分别用软件 MEGA 7进行聚类,并两端切齐。切齐后的单基因序列用软件 Mesquite v 3.1串联为多基因序列组合。采用RAXML软件构建最大似然法(Maximum likelihood)系统发育树,系统发育树的每个分支的统计学显著性分析以自展(Bootstrap)进行检验,重复次数为1 000次。
2 结果与分析
2.1 储藏稻谷真菌分离及初步鉴定
本研究对13个省储藏1年的稻谷进行了真菌分离培养和菌落计数,共获得真菌 397 株。首先采用 ITS 片段对所获得的真菌分离株进行初步鉴定(通过BLASTn在NCBI数据库上对其进行注释),将分离株初步鉴定至属水平或属内复合群水平。后续补充了 TUB基因序列,通过 ITS和TUB基因序列的比对分析结合形态观察结果,将获得的菌株初步鉴定至种水平。397个菌株分别属于2门4纲5目7科9属49种(图1)。

图1 我国粮仓储藏稻谷不同粮层真菌群落组成
Fig. 1 The fungal community composition of stored rice grains from different grain layers in warehouses of China
2.2 储藏稻谷可培养真菌群落结构组成
根据分离株初步鉴定结果分析不同真菌物种在样品中的分布情况如图1所示,曲霉属Aspergillus丰度最高,为22种,青霉属Penicillium次之,为 10种。曲霉属 Aspergillus和青霉属Penicillium数量共占物种总数的65%,且曲霉属Aspergillus的相对丰度远高于其它属。表明稻谷储藏期间的真菌主要为曲霉属 Aspergillus。本研究中,将所有样品中相对丰度大于 40%且出现 3次以上的真菌物种定为优势种,其中多育曲霉A.proliferans、蒙地曲霉 A. montevidensis、黄曲霉A. flavus和黑曲霉A. niger是我国储藏稻谷的优势菌。南北方地区的优势菌明显不同,北方以耐旱真菌多育曲霉 A. proliferans和蒙地曲霉 A.montevidensis为主,主要分布在粮堆上层。南方以蒙地曲霉A. montevidensis、黄曲霉A. flavus和黑曲霉A. niger为主,主要分布在粮堆各层。菌落计数统计结果表明,与编号S49、S46、S36及S17分离株近似的菌落数量最多。其中S49、S50、N6、N12、P54分离株疑似为蒙地曲霉 A.montevidensis;S46、S47、S52分离株疑似为多育曲霉 A. proliferans;P1、P2、P3、S36分离株疑似为黑曲霉 A. niger;S17、B63、B64、B65、B66、B67、B68、B70、F22分离株疑似为黄曲霉A. flavus。
2.3 储藏稻谷上优势真菌形态鉴定
如图2所示,分离株S49、S50、N6、N12、P54初步鉴定为蒙地曲霉 A. montevidensis,在SCA培养基上菌落生长较为迅速,正面为橄榄绿色,呈颗粒状,反面为墨绿色,在 MEA培养基上菌落生长缓慢,中央呈淡黄色,边缘为深绿色,反面为淡黄色。闭囊壳通常为黄色球形或近球形,直径为75~220 µm。分生孢子头为辐射状或疏松柱状,产孢结构单层,瓶梗为(5~12)×(2.5~3.5)µm,分生孢子球形、卵形或洋梨形,大小约为(4~6.5)×(3~5)µm,较大,表面粗糙,具小刺。分离株 46、S47、S52初步鉴定为多育曲霉 A.proliferans,在 SCA培养基上菌落质地为絮状,较厚,正反面颜色均为橄榄黄色,在 MEA培养基上不生长,在20%蔗糖CA培养基生长较为局限,形成较小的灰白色菌落,闭囊壳球形或近球形。孢梗茎壁光滑,分生孢子头直径达 70~150µm,为辐射状。顶囊为近球形,产孢结构单层,瓶梗(9~14)×(3.5~5)µm。分生孢子呈球形或近球形,大小约为(6~10)×(4~7)µm,壁粗糙,具小刺。分离株 S17、B63、B64、B65、B66、B67、B68、B70和F22初步鉴定为黄曲霉A. flavus,在CA培养基上生长迅速,菌落颜色为黄绿色或草绿色,反面为无色至淡褐色。在 MEA培养基上菌落质地丝绒状至絮状,较厚,黄绿色,菌落反面无色。孢梗茎壁厚,无色,粗糙具小刺。顶囊为近球形至烧瓶形,直径为9~65 µm。产孢结构通常为双层,梗基(6.0~19)×(3~6)µm,瓶梗(6~12)×(2.5~4)µm。分生孢子多为球形或近球形,直径2.5~6.5 µm,壁粗糙具小刺。菌核初期白色,老后呈褐黑色,球形或近球形。P1、P2、P3、S36初步鉴定为黑曲霉A. niger,在CA培养基上生长迅速,菌落表面呈暗褐黑色至炭黑色,菌落平坦或中心稍凸起,质地为致密丝绒状,菌落反面无色至淡黄色。有的菌株会产生菌核。在 MEA培养基上,菌落呈褐黑色至炭黑色,有规则或不规则的辐射状沟纹,质地丝绒状,比在 CA培养基上产生的菌丝更多,菌落反面为无色至淡黄色。孢梗茎壁光滑。分生孢子头初期为球形,老后分裂成几个圆柱状结构。顶囊球形或近球形,直径为30~80 µm。产孢结构双层,瓶梗为(7~ 12)×(2~3)µm,梗基为(10~70)×(3~14)µm,分生孢子球形或近球形,直径3~5.5 µm,壁粗糙有小刺,偶有近于光滑的孢子。
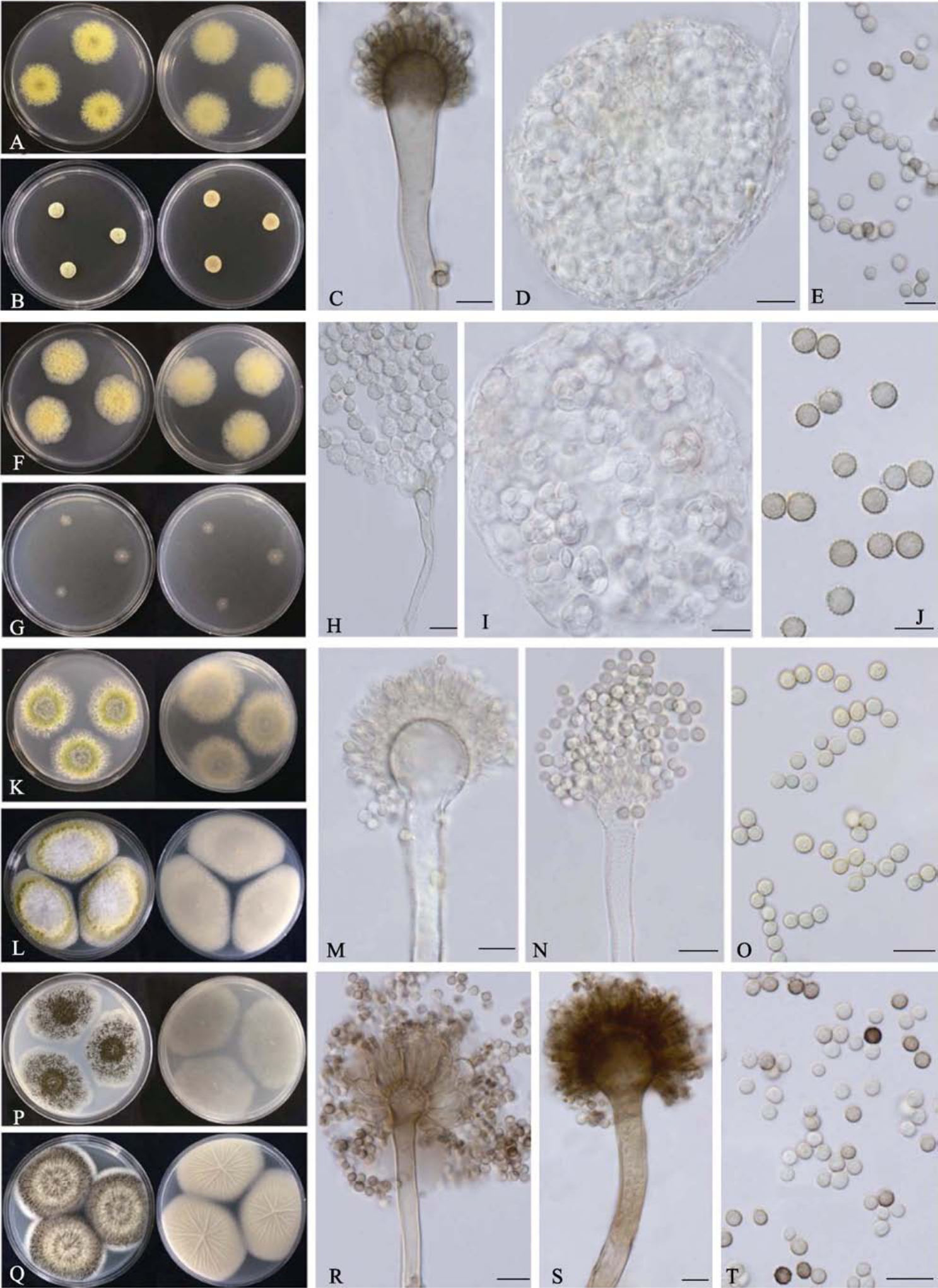
图2 优势真菌的形态结构
Fig.2 Morphological structure of dominant fungal species
注:A-E:蒙地曲霉,A(SCA,正,反),B(MEA,正,反),7d;C-E(分生孢子梗,分生孢子头,闭囊壳,子囊孢子,分生孢子);F-J多育曲霉:F(SCA,正,反),G(20%CA,正,反),7d;H-J(分生孢子梗,分生孢子头,闭囊壳,子囊孢子,分生孢子);K-O:黄曲霉:K(PDA,正,反),L(MEA,正,反),7d;M-O(分生孢子梗,分生孢子头,分生孢子);P-T:黑曲霉,P(PDA,正,反),T(MEA,正,反),7d;R-T(分生孢子梗,分生孢子头,分生孢子);标尺=10 μm。
Note: A-E: A. montevidensis, A (SCA, Upper, Lower), B (MEA, Upper, Lower), 7d; C-E (Conidiophore, Conidial head, Cleistothecia,Ascospore, Conidia); A. proliferans: F (SCA, Upper, Lower), G (20%CA, Upper, Lower), 7d; H-J (Conidiophore, Conidial head,Cleistothecia, Ascospore, Conidia); K-O: A. flavus, K (PDA, Upper, Lower), L (MEA, Upper, Lower),7d; M-O (Conidiophore, Conidial head,Conidia); K-O: A. niger, K (PDA, Upper, Lower), L (MEA, Upper, Lower),7d; M-O (Conidiophore, Conidial head, Conidia); Scales=10 μm.
2.4 储藏稻谷上优势真菌分子生物学鉴定
经过ITS比对分析和形态特征鉴定,发现储藏稻谷上的优势菌主要属于3个类群,第一类属于曲霉属Aspergillus中耐旱真菌类群(早期属于Eurotium散囊菌属),具有嗜高渗透压的特性(耐旱性);第二类属于黄色组 Aspergillus section Flavi;第三类属于黑色组 Aspergillus section Nigri。对同一组内的真菌物种而言,ITS序列在鉴定过程中高度保守,与其物种的形态特征相近,区分困难,容易混淆。因此,选择真菌的 ITS、TUB、CAM和RPB2基因进行测序,结合同类群内近似物种的模式菌株和国际权威菌株序列进行多基因系统发育分析,最终确定真菌的鉴定结果。
2.4.1 储藏稻谷中耐旱真菌类群的多基因系统发育分析
基于四个基因片段的多位点系统发育分析方法,推测曲霉属Aspergillus中耐旱真菌物种之间的关系(图3)。最终得到的比对数据集包含 21个分离菌株和28个标准菌株(附表1),包括2 283个碱基(ITS为500,TUB为411,CAM为429和RPB2为943,包括比对间隙),菌株 Penicillium chrysogenum CBS 30648 作为外群。结果表明,分离株 N13、N21、N24、P47、P48、P51、P52、P55、P56和S55所测序列与谢瓦曲霉A. chevalieri标准序列位于系统发育树一个分支上;S49、S50、N6、N12、P54所测序列与蒙地曲霉 A.montevidensis标准序列位于系统发育树一个分支上;S46、S47、S52所测序列与多育曲霉 A.proliferans标准序列位于系统发育树一个分支上。最终确定储藏稻谷中属于耐旱真菌的优势种为蒙地曲霉A. montevidensis、谢瓦曲霉 A. chevalieri和多育曲霉A. proliferans。本研究相关分离株的基因序列已经提交至 NCBI数据库,登录号见附表1。

图3 基于多基因序列(ITS、TUB、CAM、RPB2)构建的曲霉属中的部分曲霉(耐旱真菌)物种的系统发育树
Fig.3 Phylogenetic tree of Aspergillus section Aspergillus (Xerophilic fungi) inferred from combined ITS, TUB, CAM and RPB2
注:分支上的数字表示自展支持率,只显示自展支持率大于50%的值。
Note: Numbers above branches are bootstrap values. Only values above 50% are showed.
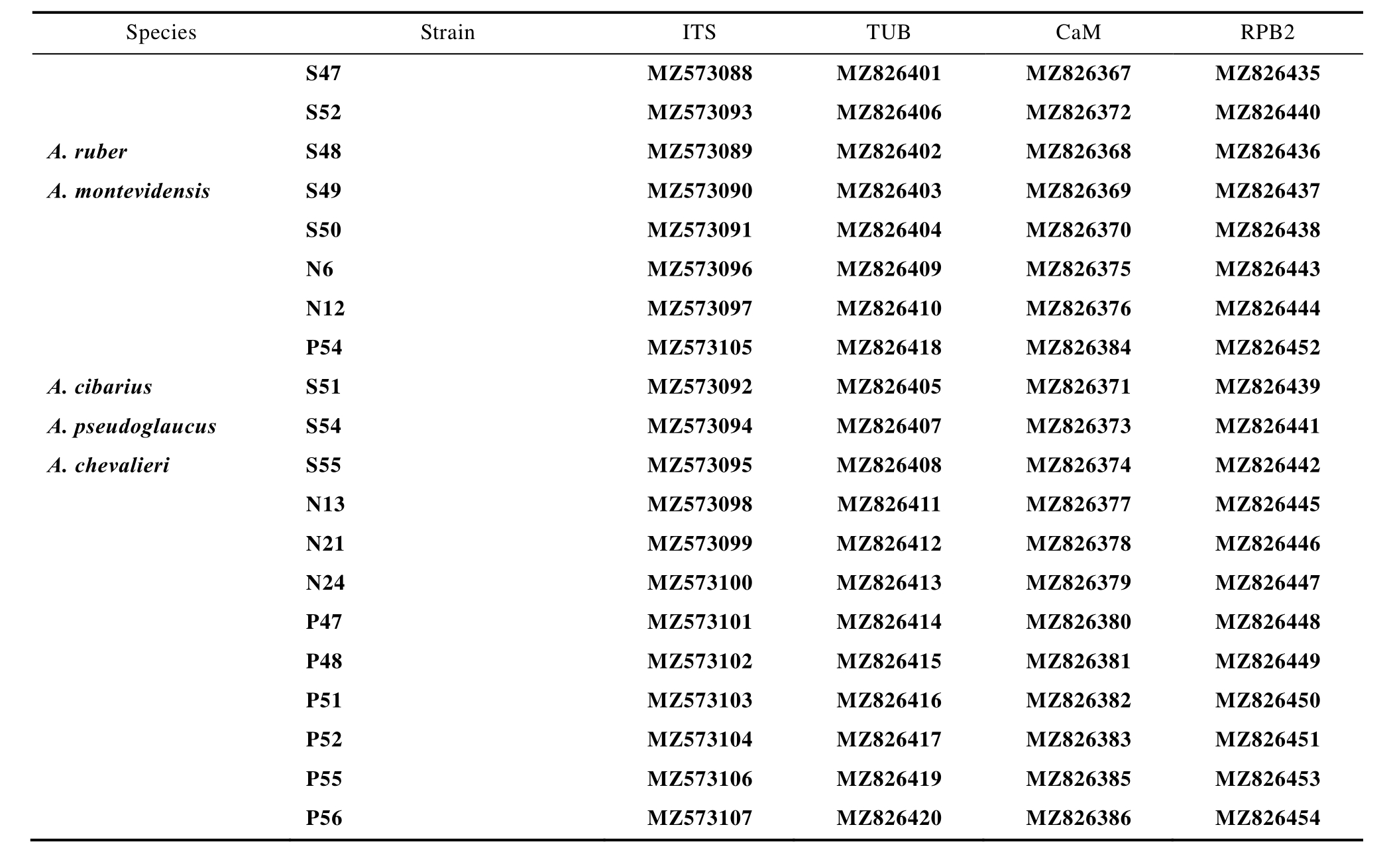
附表1 本实验分离菌株及标准分离株在GenBank登录号
Attached table 1 Standard isolates used in this study and their GenBank accession numbers

Species Strain ITS TUB CaM RPB2 A. chevalieri NRRL 79 EF652069 EF651912 EF652003 EF651955 NRRL 4755 EF652071 EF651913 EF652004 EF651956 CBS 141769 LT670927 LT671023 LT671024 LT671025 A. cibarius KACC 49766 LT670930 LT671032 LT671033 LT671034 CGMCC 3.06498 LT670934 LT671046 LT671047 LT671048 CCF 5383 LT670932 LT671040 LT671041 LT671042 A. aurantiacoflavus CBS 141930 LT670917 LT670993 LT670994 LT670995 A. costiformi CBS 101749 HE615136 HE801338 HE801320 HE801309 CGMCC 3.06520 LT670937 LT671055 LT671056 LT671057 A. cristatus NRRL 4222 NR135341 EF651914 EF652001 EF651957 A. glaucus NRRL 116 NR135337 EF651887 EF651989 EF651934 CCF 5381 LT670942 LT671070 LT671071 LT671072 A. intermedius NRRL 82 NR137448 EF651892 EF652012 EF651958 CGMCC 3.03968 LT670946 LT671082 LT671083 LT671084 A. montevidensis NRRL 108 NR137449 EF651898 EF652020 EF651964 A. amstelodami NRRL 4716 EF652079 EF651899 EF652018 EF651965 A. hollandicus NRRL 25850 EF652082 EF651900 EF652021 EF651966 A. montevidensis CGMCC 3.03888 LT670954 LT671106 LT671107 LT671108 A. proliferans NRRL 1908 NR135339 EF651891 EF651988 EF651941 CCF 4115 FR851850 FR851855 HE578090 HE578107 CCF 4232 HE615129 HE801329 HE801317 HE801306 A. pseudoglaucus NRRL 40 NR135336 EF651917 EF652007 EF651952 NRRL 25865 EF652065 EF651918 EF652008 EF651953 CCF 4011 FR839678 FR775358 FR751446 HE578111 A. ruber NRRL 52 NR131286 EF651920 EF652009 EF651947 CCF 2920 FR727112 FR775357 FR751444 HE974430 CGMCC 3.00457 LT670980 LT671187 LT671188 LT671189 A. zutongqii CBS 141773 LT670986 LT671206 LT671207 LT671208 A. proliferans S46 MZ573087 MZ826400 MZ826366 MZ826434
续附表1
S p e c i e s S t r a i n I T S T U B C a M R P B 2 S 4 7 M Z 5 7 3 0 8 8 M Z 8 2 6 4 0 1 M Z 8 2 6 3 6 7 M Z 8 2 6 4 3 5 S 5 2 M Z 5 7 3 0 9 3 M Z 8 2 6 4 0 6 M Z 8 2 6 3 7 2 M Z 8 2 6 4 4 0 A. r u b e r S 4 8 M Z 5 7 3 0 8 9 M Z 8 2 6 4 0 2 M Z 8 2 6 3 6 8 M Z 8 2 6 4 3 6 A. m o n t e v i d e n s i s S 4 9 M Z 5 7 3 0 9 0 M Z 8 2 6 4 0 3 M Z 8 2 6 3 6 9 M Z 8 2 6 4 3 7 S 5 0 M Z 5 7 3 0 9 1 M Z 8 2 6 4 0 4 M Z 8 2 6 3 7 0 M Z 8 2 6 4 3 8 N 6 M Z 5 7 3 0 9 6 M Z 8 2 6 4 0 9 M Z 8 2 6 3 7 5 M Z 8 2 6 4 4 3 N 1 2 M Z 5 7 3 0 9 7 M Z 8 2 6 4 1 0 M Z 8 2 6 3 7 6 M Z 8 2 6 4 4 4 P 5 4 M Z 5 7 3 1 0 5 M Z 8 2 6 4 1 8 M Z 8 2 6 3 8 4 M Z 8 2 6 4 5 2 A. c i b a r i u s S 5 1 M Z 5 7 3 0 9 2 M Z 8 2 6 4 0 5 M Z 8 2 6 3 7 1 M Z 8 2 6 4 3 9 A. p s e u d o g l a u c u s S 5 4 M Z 5 7 3 0 9 4 M Z 8 2 6 4 0 7 M Z 8 2 6 3 7 3 M Z 8 2 6 4 4 1 A. c h e v a l i e r i S 5 5 M Z 5 7 3 0 9 5 M Z 8 2 6 4 0 8 M Z 8 2 6 3 7 4 M Z 8 2 6 4 4 2 N 1 3 M Z 5 7 3 0 9 8 M Z 8 2 6 4 1 1 M Z 8 2 6 3 7 7 M Z 8 2 6 4 4 5 N 2 1 M Z 5 7 3 0 9 9 M Z 8 2 6 4 1 2 M Z 8 2 6 3 7 8 M Z 8 2 6 4 4 6 N 2 4 M Z 5 7 3 1 0 0 M Z 8 2 6 4 1 3 M Z 8 2 6 3 7 9 M Z 8 2 6 4 4 7 P 4 7 M Z 5 7 3 1 0 1 M Z 8 2 6 4 1 4 M Z 8 2 6 3 8 0 M Z 8 2 6 4 4 8 P 4 8 M Z 5 7 3 1 0 2 M Z 8 2 6 4 1 5 M Z 8 2 6 3 8 1 M Z 8 2 6 4 4 9 P 5 1 M Z 5 7 3 1 0 3 M Z 8 2 6 4 1 6 M Z 8 2 6 3 8 2 M Z 8 2 6 4 5 0 P 5 2 M Z 5 7 3 1 0 4 M Z 8 2 6 4 1 7 M Z 8 2 6 3 8 3 M Z 8 2 6 4 5 1 P 5 5 M Z 5 7 3 1 0 6 M Z 8 2 6 4 1 9 M Z 8 2 6 3 8 5 M Z 8 2 6 4 5 3 P 5 6 M Z 5 7 3 1 0 7 M Z 8 2 6 4 2 0 M Z 8 2 6 3 8 6 M Z 8 2 6 4 5 4
2.4.2 储藏稻谷上黄曲霉复合群内物种的多基因系统发育分析
基于四个基因片段的多位点系统发育分析方法,推测稻谷上黄曲霉复合群Aspergillus section Flavi内物种之间的关系(图4)。最终得到的比对数据集包含9个分离菌株和21个标准菌株(附表2),包括2 429个碱基(ITS为542,TUB为509,CAM为460和RPB2为918,包括比对间隙),菌株P. chrysogenum CBS 30648作为外群。S17、B63、B64、B65、B66、B67、B68、B70 和 F22所测序列与黄曲霉A. flavus和米曲霉A. oryzae标准序列位于系统发育树一个分支上(图4)。表明上述分离株可能为黄曲霉 A. flavus或米曲霉 A.oryzae。通过这 4个基因片段的多基因系统发育分析无法将黄曲霉A. flavus和米曲霉A. oryzae进行区分。结合2.1中优势菌的形态学鉴定的结果,可将分离株准确鉴定为黄曲霉 A. flavus。本研究分离株的基因序列已经提交至 NCBI数据库,登录号见附表2。

图4 基于多基因序列(ITS、TUB、CAM、RPB2)构建的黄曲霉复合群物种的系统发育树
Fig.4 Phylogenetic tree of Aspergillus section Flavi inferred from combined ITS, TUB, CAM and RPB2
注:分支上的数字表示自展支持率,只显示自展支持率大于50%的值。
Note: Numbers above branches are bootstrap values. Only values above 50 % are showed.
附表2 本实验分离菌株及标准分离株在GenBank登录号
Attached table 2 Standard isolates used in this study and their GenBank accession numbers
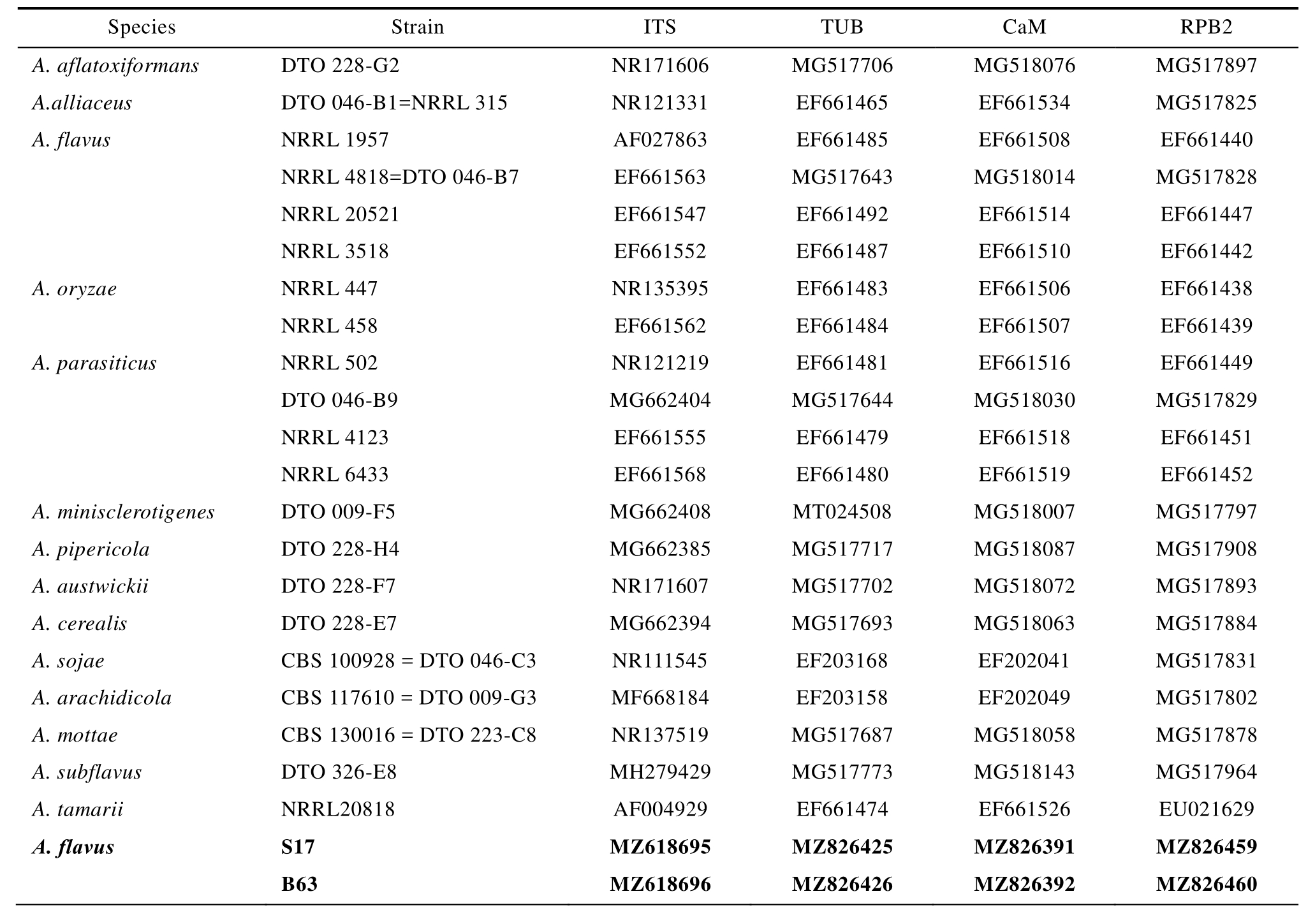
Species Strain ITS TUB CaM RPB2 A. aflatoxiformans DTO 228-G2 NR171606 MG517706 MG518076 MG517897 A.alliaceus DTO 046-B1=NRRL 315 NR121331 EF661465 EF661534 MG517825 A. flavus NRRL 1957 AF027863 EF661485 EF661508 EF661440 NRRL 4818=DTO 046-B7 EF661563 MG517643 MG518014 MG517828 NRRL 20521 EF661547 EF661492 EF661514 EF661447 NRRL 3518 EF661552 EF661487 EF661510 EF661442 A. oryzae NRRL 447 NR135395 EF661483 EF661506 EF661438 NRRL 458 EF661562 EF661484 EF661507 EF661439 A. parasiticus NRRL 502 NR121219 EF661481 EF661516 EF661449 DTO 046-B9 MG662404 MG517644 MG518030 MG517829 NRRL 4123 EF661555 EF661479 EF661518 EF661451 NRRL 6433 EF661568 EF661480 EF661519 EF661452 A. minisclerotigenes DTO 009-F5 MG662408 MT024508 MG518007 MG517797 A. pipericola DTO 228-H4 MG662385 MG517717 MG518087 MG517908 A. austwickii DTO 228-F7 NR171607 MG517702 MG518072 MG517893 A. cerealis DTO 228-E7 MG662394 MG517693 MG518063 MG517884 A. sojae CBS 100928 = DTO 046-C3 NR111545 EF203168 EF202041 MG517831 A. arachidicola CBS 117610 = DTO 009-G3 MF668184 EF203158 EF202049 MG517802 A. mottae CBS 130016 = DTO 223-C8 NR137519 MG517687 MG518058 MG517878 A. subflavus DTO 326-E8 MH279429 MG517773 MG518143 MG517964 A. tamarii NRRL20818 AF004929 EF661474 EF661526 EU021629 A. flavus S17 MZ618695 MZ826425 MZ826391 MZ826459 B63 MZ618696 MZ826426 MZ826392 MZ826460
续附表2

Species Strain ITS TUB CaM RPB2 B64 MZ618697 MZ826427 MZ826393 MZ826461 B65 MZ618698 MZ826428 MZ826394 MZ826462 B66 MZ618699 MZ826429 MZ826395 MZ826463 B67 MZ618700 MZ826430 MZ826396 MZ826464 B68 MZ618701 MZ826431 MZ826397 MZ826465 B70 MZ618702 MZ826432 MZ826398 MZ826466 F22 MZ618703 MZ826433 MZ826399 MZ826467
2.4.3 储藏稻谷上黑曲霉复合群内物种的多基因系统发育分析
基于四个基因片段的多位点系统发育分析方法,推测稻谷上黑曲霉复合群Aspergillus section Nigri内物种之间的关系(图5)。最终得到的比对数据集包含4个分离菌株和14个标准菌株(附表3),包括2 002个碱基(ITS为448,TUB为414,CAM为450和RPB2为690,包括比对间隙),菌株P. chrysogenum CBS 30648作为外群。实验室分离菌株 P1、P2、P3、S36所测序列与黑曲霉A. niger标准序列位于系统发育树一个分支上。最终确定上述分离株均为黑曲霉A. niger。本研究分离株的基因序列已经提交至 NCBI数据库,登录号见附表3。

图5 基于多基因序列(ITS、TUB、CAM、RPB2)构建的黑曲霉复合群物种的系统发育树
Fig.5 Phylogenetic tree of Aspergillus section Nigri inferred from combined ITS, TUB, CAM and RPB2
注:分支上的数字表示自展支持率,只显示自展支持率大于50%的值。
Note: Numbers above branches are bootstrap values. Only values above 50% are showed.
附表3 本实验分离菌株及标准分离株在GenBank登录号
Attached table 3 Standard isolates used in this study and their GenBank accession numbers

Species Strain ITS TUB CaM RPB2 A. neoniger CBS 115656 FJ491682 FJ491691 FJ491700 —A. niger ATCC 16888 NR111348 KU897009 — —ITEM 4501 = CBS 554.65 AY585554 AY585536 AJ964872 —ATHUM 5044 EU982008 EU982067 — EU982093 ATHUM 2539 EU982009 EU982068 — EU982092 A. costaricaensis CBS 115574 NR103604 AY820014 EU163268 —A. awamori CBS 557.65=NRRL 4948 KF288139 KF288129 KF288119 —A. vadensis CBS 113365 MH862923 GU296695 FN594560 —A. eucalypticola CBS 122712=53A2 EU482439 EU482435 EU482433 MN969070 A. piperis CBS 112811 NR077191 AY820013 FN594554 —A. welwitschiae CBS 139.54 NR137513 MN969369 LC570775 MN969100 A. brasiliensis CBS 101740 AJ280010 AY820006 AM295175 —A. carbonarius CBS 111.26 DQ900605 AY585532 AJ964873 —A. sclerotioniger CBS 115572 DQ900606 AY819996 EU163271 —A. niger P1 MZ618691 MZ826421 MZ826387 MZ826455 P2 MZ618692 MZ826422 MZ826388 MZ826456 P3 MZ618693 MZ826423 MZ826389 MZ826457 S36 MZ618694 MZ826424 MZ826390 MZ826458
3 讨论和结论
本研究对我国 13个省份储藏期稻谷进行了真菌分离、菌落计数和鉴定,最终获得397株菌,其中21个分离株代表储藏期稻谷上优势种,经形态学观察和多基因系统发育分析,将21个分离株准确鉴定为4个种,分别为黄曲霉A. flavus、黑曲霉A. niger、蒙地曲霉A. montevidensis和多育曲霉A. proliferans。这与已报道的储藏期粮食上常见的优势真菌种物略有差异[15-16]。其中黄曲霉A. flavus和黑曲霉A. niger在我国南方储藏稻谷中普遍存在[7,17],当优势种为黄曲霉 A. flavus和黑曲霉A. niger时,黄曲霉毒素和赭曲霉毒素污染的风险可能增加[18],这两种真菌是粮食行业需重点关注的危害真菌。很多研究中提到A. glaucus灰绿曲霉是储粮上常见的优势菌[11],我们分离的优势菌蒙地曲霉A. montevidensis原命名为阿姆斯特丹曲霉A. amstelodami,经Chen等[14]系统研究后,更名为蒙地曲霉 A. montevidensis,属Raper等(1965)分类系统中灰绿曲霉群A. glaucus group中典型代表,经真菌命名溯源,与前人研究是一致的。而多育曲霉A. proliferans属于早期研究中提到的局限曲霉群A. restrictus group,也是储粮上常见真菌物种之一[14]。这些物种具有嗜高渗透压的特性(耐旱性),能够在水分活度(aW)低于0.75条件下生长[19],通常生长缓慢,但若不采取防控措施,长期生长仍会对稻谷品质造成影响,如导致发芽率降低,脂肪酸值升高,出现色泽变暗,气味异常等情况,因此作为粮堆霉变发热早期监测预警主要指示菌群[20]。
粮食中真菌种、属的鉴定,由于技术条件和研究方法不同,且真菌形态具有多样性,使得粮食真菌精准鉴定方面还存在一些不足,如粮食上耐旱真菌的鉴定结果和种名一直存在争议,黄曲霉A. flavus和米曲霉A. oryzae很难准确区分,不利于后期研究学者进行深入研究。本研究采用形态学观察及ITS、TUB、CAM和RPB2多基因系统发育分析相结合的方法,对储藏稻谷上曲霉属Aspergillus的3个主要优势类群进行了深入研究,并给出了主要优势种的精准鉴定方法,可为粮食中基因型或表型相似真菌物种的准确鉴定提供参考。
[1]张瑛, 吴先山, 吴敬德, 等. 稻谷储藏过程中理化特性变化的研究[J]. 中国粮油学报, 2003, 18 (6): 20-24.ZHANG Y, WU X S, WU J D, et al. The effects of natural fermentation on characteristics of the rice starch granule[J].Journal of the Chinese Cereals and Oils Association, 2003, 18(6): 20-24.
[2]GONCALVES A, GKRILLAS A, DORNE J L, et al. Pre- and postharvest strategies to minimize mycotoxin contamination in the rice food chain[J]. Comprehensive Reviews in Food Science and Food Safety, 2019, 18(2): 441-454.
[3]张海洋, 欧阳毅, 祁智慧, 等. 稻谷储存水分和温度对真菌生长和稻谷主要品质的影响[J]. 粮油食品科技, 2017, 25 (2):39-43.ZHANG H Y, OUYANG Y, QI Z H, et al. Influence of storage moisture continent and temperature on green of fungi and quality of stored paddy[J]. Science and Technology of Cereals, Oils and Foods, 2017, 25 (2): 39-43.
[4]ZHAO Y, WANG Q, HUANG J, et al. Mycotoxin contamination and presence of mycobiota in rice sold for human consumption in China[J]. Food Control, 2019, 98: 19-23.
[5]REDDY O R, SATHYANARAYANA N. Seed-borne fungi of rice and quarantine significance[M]. Springer Netherlands, 2001.
[6]ZULKIFLI N A, ZAKARIA L. Morphological and molecular diversity of Aspergillus from corn grain used as livestock feed[J]. Journal of Biosciences, 2017, 24 (1): 26-34.
[7]葛志文, 方宝庆, 邱伟芬, 等. 储藏稻谷优势霉菌的分离鉴定及特征分析[J]. 粮油食品科技, 2019, 27 (3): 72-77.GE Z W, FANG B Q, QIU W F, et al. Separation, identification and characteristics analysis of dominant mould in stored rice [J].Science and Technology of Cereals, Oils and Foods, 2019, 27(3): 72-77.
[8]YAZDANI D, ZAINAL ABIDIN M A, TAN Y H, et al.Molecular identification of Aspergillus and Eurotium species isolated from rice and their toxin-producing ability[J].Microbiology, 2011, 80 (5): 720-727.
[9]吴红萍, 翟世博, 杜晨辉, 等. 海南省储粮稻谷的霉菌多样性分析[J]. 贵州农业科学, 2015, 43 (4): 138-141.WU H P, ZHAI S B, DU C H, et al. Analysis of mold diversity of grain storage paddy in Hainan Province[J]. Guizhou Agricultural Sciences, 2015, 43 (4):138-141.
[10]都立辉, 和肖营, 刘凌平, 等. 淮稻5号的真菌多样性及其储藏过程中可培养的优势真菌[J]. 中国农业科学, 2016, 49 (7):1371-1381.DU L H, HE X Y, LIU L P, et al. Fungal diversity of Huaidao No. 5 rice and the dominant culturable fungal strains during storage[J]. Scientia Agricultura Sinica, 2016, 49 (7): 1371-1381.
[11]李娜, 周红丽, 张兵, 等. 稻谷霉菌的分离鉴定及其对稻谷品质的影响研究[J]. 中国粮油学报, 2020, 35 (12): 115-124.LI N, ZHOU H L, ZHANG B, et al. Isolation and identification of rice mold and its effect on rice quality[J]. Journal of the Chinese Cereals and Oils Association, 2020, 35 (12): 115-124.
[12]SAMSON R A, VISAGIE C M, HOUBRAKEN J, et al.Phylogeny, identification and nomenclature of the genus Aspergillus[J]. Studies in mycology, 2014, 78 (78): 141-173.
[13]FRISVAD J C, HUBKA V, EZEKIEL C N, et al. Taxonomy of Aspergillus section Flavi and their production of aflatoxins,ochratoxins and other mycotoxins[J]. Studies in Mycology, 2019,93: 1-63.
[14]CHEN A J, HUBKA V, FRISVAD J C, et al. Polyphasic taxonomy of Aspergillus section Aspergillus (formerly Eurotium), and its occurrence in indoor environments and food[J]. Studies in Mycology, 2017, 88: 37-135.
[15]KUMARI R, JAYACHANDRAN L E, GHOSH A K.Investigation of diversity and dominance of fungal biota in stored wheat grains from governmental warehouses in West Bengal, India[J]. J Sci Food Agric, 2019, 99 (7): 3490-3500.
[16]OH J Y, SANG M K, OH J E, et al. Microbial population,aflatoxin contamination and predominant Aspergillus species in Korean stored rice[J]. The Plant Pathology Journal, 2010, 26 (2):121-129.
[17]祁智慧, 周欣, 张海洋, 等. 中国稻谷可培养真菌群落组成及多样性[J]. 菌物学报, 2020, 39 (4): 683-694.QI Z H, ZHOU X, ZHANG H Y, et al. Community composition and diversity of culturable fungi from the rice grains in China[J].Mycosystema, 2020, 39 (4): 683-694.
[18]BARANYI N, MUTHUSAMY C, VAGVOLGYI C, et al.Mycotoxin producers in the Aspergillus genus: An update[J].Acta Biologica Szegediensis, 2015, 59 (2): 151-167.
[19]FLEURAT-LESSARD F. Integrated management of the risks of stored grain spoilage by seedborne fungi and contamination by storage mould mycotoxins – An update[J]. Journal of Stored Products Research, 2017, 71: 22-40.
[20]GRECO M, KEMPPAINEN M, POSE G, et al. Taxonomic characterization and secondary metabolite profiling of Aspergillus Section Aspergillus contaminating feeds and feedstuffs[J]. Toxins, 2015, 7 (9): 3512-3537.
备注:本文的彩色图表可从本刊官网(http//lyspkj.ijournal.cn)、中国知网、万方、维普、超星等数据库下载获取。